Հոմոլոգ ռեկոմբինացիա

Հոմոլոգ ռեկոմբինացիա, գենետիկ ռեկոմբինացիայի տեսակ, որի ժամանակ գենետիկ տեղեկատվությունը փոխանակվում է երկշղթա կամ միաշղթա նուկլեինաթթուների երկու նմանատիպ կամ նույնական մոլեկուլների միջև (սովորաբար ԴՆԹ, ինչպես բջջային օրգանիզմներում, բայց կարող է լինել նաև ՌՆԹ վիրուսներում):
Հոմոլոգ ռեկոմբինացիան լայնորեն օգտագործվում է բջիջների կողմից՝ ճշգրիտ կերպով վերականգնելու ԴՆԹ-ի վնասակար ճեղքերը, որոնք տեղի են ունենում ԴՆԹ-ի երկու շղթաների վրա, որոնք հայտնի են որպես կրկնակի շղթաների ճեղքեր (DSB), մի գործընթացում, որը կոչվում է հոմոլոգ ռեկոմբինացիոն վերականգնում (HRR)[1]:
Հոմոլոգ ռեկոմբինացիան նաև առաջացնում է ԴՆԹ-ի հաջորդականությունների նոր համակցություններ մեյոզի ընթացքում, գործընթաց, որով էուկարիոտները ստեղծում են գամետային բջիջներ, ինչպես կենդանիների սերմնաբջիջը և ձվաբջիջը: ԴՆԹ-ի այս նոր համակցությունները ներկայացնում են սերունդների գենետիկական փոփոխությունը, որն իր հերթին հնարավորություն է տալիս պոպուլյացիաներին հարմարվել էվոլյուցիայի ընթացքում[2]:
Հոմոլոգ ռեկոմբինացիան օգտագործվում է նաև հորիզոնական գեների փոխանցման մեջ՝ բակտերիաների և վիրուսների տարբեր շտամների և տեսակների միջև գենետիկ նյութի փոխանակման համար: Հորիզոնական գեների փոխանցումը բակտերիաների մեջ հակաբիոտիկների դիմադրության տարածման առաջնային մեխանիզմն է:
Չնայած հոմոլոգ ռեկոմբինացիան լայնորեն տարբերվում է տարբեր օրգանիզմների և բջիջների տեսակների միջև, երկշղթա ԴՆԹ-ի (dsDNA) համար ձևերի մեծ մասը ներառում է նույն հիմնական քայլերը: Կրկնակի շղթայի ընդհատումից հետո ԴՆԹ-ի հատվածները կտրվածքի 5' ծայրերի շուրջը կտրվում են մի գործընթացով, որը կոչվում է ռեզեկցիա: Հետևյալ շղթայի ներխուժման քայլում կոտրված ԴՆԹ-ի մոլեկուլի 3' ծայրը «ներխուժում» է նմանատիպ կամ նույնական ԴՆԹ մոլեկուլ, որը կտրված չէ: Թելերի ներխուժումից հետո իրադարձությունների հետագա հաջորդականությունը կարող է հետևել ստորև քննարկված երկու հիմնական ուղիներից որևէ մեկին, DSBR (կրկնակի շղթայի ճեղքի վերականգնում) ուղին կամ SDSA (սինթեզից կախված շղթայի ձուլման) ուղին: ԴՆԹ-ի վերանորոգման ժամանակ տեղի ունեցող հոմոլոգ ռեկոմբինացիան հանգեցնում է ոչ խաչաձև արտադրանքի, որն իրականում վերականգնում է վնասված ԴՆԹ մոլեկուլը, ինչպես այն կար մինչև կրկնակի շղթայի վնասվելը:
Հոմոլոգ ռեկոմբինացիան պահպանված է կյանքի բոլոր երեք տիրույթներում, ինչպես նաև ԴՆԹ և ՌՆԹ վիրուսներում, ինչը ենթադրում է, որ այն գրեթե ունիվերսալ կենսաբանական մեխանիզմ է: Պրոտիստների՝ էուկարիոտ միկրոօրգանիզմների բազմազան խմբի՝ հոմոլոգ ռեկոմբինացիա գեների հայտնաբերումը մեկնաբանվել է որպես ապացույց, որ հոմոլոգ ռեկոմբինացիան առաջացել է էուկարիոտների էվոլյուցիայի սկզբում։ Քանի որ դրանց դիսֆունկցիան խստորեն կապված է քաղցկեղի մի քանի տեսակների նկատմամբ զգայունության բարձրացման հետ, այն սպիտակուցները, որոնք հեշտացնում են հոմոլոգ ռեկոմբինացիան, ակտիվ հետազոտության թեմաներ են: Հոմոլոգ ռեկոմբինացիան օգտագործվում է նաև գեների թիրախավորման մեջ՝ նպատակային օրգանիզմների մեջ գենետիկ փոփոխություններ մտցնելու տեխնիկա: 2007 թվականին այս տեխնիկայի զարգացման համար Մարիո Կապեկին, Մարտին Էվանսը և Օլիվեր Սմիթիսը արժանացել են ֆիզիոլոգիայի և բժշկության Նոբելյան մրցանակի։ Կապեկին[3] և Սմիթիզը[4] ինքնուրույն կիրառություն են գտել մկների սաղմնային ցողունային բջիջների համար, սակայն DSB-ի վերականգնման մոդելի հիմքում ընկած խիստ պահպանված մեխանիզմները, ներառյալ փոխակերպված ԴՆԹ-ի միատեսակ հոմոլոգ ինտեգրումը (գենային թերապիա), առաջին անգամ ցուցադրվել են Օր-Ուիվերի, Սզոստակի և Ռոտշտեինի կողմից պլազմիդային փորձերում[5][6][7]: 1970-1980-ական թվականներին պլազմիդից առաջացած DSB-ի ուսումնասիրությունը, օգտագործելով γ-ճառագայթումը[8], հանգեցրեց ավելի ուշ փորձերի՝ օգտագործելով էնդոնուկլեազներ (օրինակ՝ I-SceI)՝ կաթնասունների բջիջների գենետիկական ինժեներիայի համար քրոմոսոմները կտրելու համար, որտեղ ոչ հոմոլոգիական ռեկոմբինացիան ավելի հաճախ է տեղի ունենում, քան՝ խմորասնկերում[9]։
Պատմություն և բացահայտում
[խմբագրել | խմբագրել կոդը]
1900-ական թվականների սկզբին Ուիլյամ Բեյթսոնը և Ռեջինալդ Փանեթը բացառություն գտան ժառանգության սկզբունքներից մեկից, որն ի սկզբանե նկարագրված էր Գրեգոր Մենդելի կողմից 1860-ականներին: Ի տարբերություն Մենդելի այն մտքի, որ հատկությունները տարբերվում են ծնողից երեխային փոխանցելիս, օրինակ, որ կատվի մազերի գույնը և նրա պոչի երկարությունը ժառանգվում են միմյանցից անկախ, Բեյթսոնը և Փանեթը ցույց տվեցին, որ ֆիզիկական հատկությունների հետ կապված որոշ գեներ կարող են ժառանգվել միասին, կամ գենետիկորեն կապված[10][11]։ 1911թվականին Թոմաս Հանթ Մորգանը նկատեց, որ կապակցված հատկությունները երբեմն կարող են ժառանգվել առանձին-առանձին, առաջարկեց, որ «խաչաձևերը» կարող են առաջանալ կապված գեների միջև[12], որտեղ կապակցված գեներից մեկը ֆիզիկապես անցնում է մեկ այլ քրոմոսոմ: Երկու տասնամյակ անց Բարբարա ՄակՔլինթոկը և Հարիետ Քրեյթոնը ցույց տվեցին, որ քրոմոսոմային տրամախաչումը տեղի է ունենում մեյոզի ժամանակ[13][14], բջիջների բաժանման գործընթացում, որով ստեղծվում են սերմնաբջիջները և ձվաբջիջները: ՄակՔլինթոքի հայտնագործության հետ նույն տարում Քերթ Սթերնը ցույց տվեց, որ տրամախաչումը, որը հետագայում կոչվեց «ռեկոմբինացիա», կարող է տեղի ունենալ նաև սոմատիկ բջիջներում, ինչպիսիք են արյան սպիտակ բջիջները և մաշկի բջիջները, որոնք բաժանվում են միտոզով[13][15]:
1947 թվականին մանրէաբան Ջոշուա Լեդերբերգը ցույց տվեց, որ բակտերիաները, որոնք ենթադրվում էր, որ վերարտադրվում են միայն անսեռ ճանապարհով երկուական տրոհման միջոցով, կարող են գենետիկ ռեկոմբինացվել, որն ավելի շատ նման է սեռական վերարտադրությանը։ Այս աշխատանքը հաստատեց E. coli-ն որպես գենետիկայի մոդելային օրգանիզմ[16] և օգնեց Լեդերբերգին նվաճել 1958 թվականին Նոբելյան մրցանակը ֆիզիոլոգիայի կամ բժշկության բնագավառում[17]։ Հիմնվելով սնկերի վրա ուսումնասիրությունների վրա՝ 1964 թվականին Ռոբին Հոլիդեյն առաջարկեց մեյոզի վերահամակցման մոդել, որը ներկայացրեց հիմնական մանրամասները, թե ինչպես կարող է այդ գործընթացը աշխատել, ներառյալ նյութի փոխանակումը քրոմոսոմների միջև Հոլիդեյի հանգույցների միջոցով[18]: 1983 թվականին Ջեք Սզոստակը և գործընկերները ներկայացրեցին մոդել, որն այժմ հայտնի է որպես DSBR ուղի, որը հաշվի է առնում այն դիտարկումները, որոնք չեն բացատրվում Հոլիդեյի մոդելով[18][7]: Հաջորդ տասնամյակի ընթացքում Drosophila ճանճի, բողբոջող խմորասնկի և կաթնասունների բջիջների վրա կատարվող փորձերը հանգեցրին հոմոլոգ ռեկոմբինացիայի այլ մոդելների առաջացմանը, որոնք կոչվում են SDSA ուղիներ, որոնք միշտ չէ, որ հիմնվում են Հոլիդեյի հանգույցների վրա[18]:
Գործընթացում ներգրավված սպիտակուցների բացահայտման և դրանց մեխանիզմների որոշման հետագա աշխատանքների մեծ մասը կատարվել է մի շարք անհատների կողմից, այդ թվում՝ Ջեյմս Հաբերը, Պատրիկ Սունգը, Սթիվեն Կովալչիկովսկին և այլք:
Կորիզավորներում
[խմբագրել | խմբագրել կոդը]Հոմոլոգ ռեկոմբինացիան կարևոր է էուկարիոտների բջիջների բաժանման համար, ինչպիսիք են բույսերը, կենդանիները, սնկերը և պրոտիստները: Հոմոլոգ ռեկոմբինացիան վերականգնում է ԴՆԹ-ի կրկնակի շղթայի ճեղքերը, որոնք առաջացել են իոնացնող ճառագայթման կամ ԴՆԹ-ն վնասող քիմիական նյութերի հետևանքով[19]։ Եթե չվերականգնվեն, այս կրկնակի շղթայի ճեղքերը կարող են առաջացնել սոմատիկ բջիջների քրոմոսոմների լայնածավալ վերադասավորում[20], որն իր հերթին կարող է հանգեցնել քաղցկեղի[21]:
Ի հավելումն ԴՆԹ-ի վերականգնմանը, հոմոլոգ ռեկոմբինացիան նաև օգնում է առաջացնել գենետիկական բազմազանություն, երբ բջիջները մեյոզի ընթացքում բաժանվում են՝ դառնալով մասնագիտացված գամետային բջիջներ՝ կենդանիների մեջ սերմնաբջիջներ կամ ձվաբջիջներ, բույսերի ծաղկափոշին կամ ձվաբջջները, և սնկերի սպորները: Այն դա անում է հեշտացնելով քրոմոսոմային տրամախաչումը, որտեղ համանման, բայց ոչ նույնական ԴՆԹ-ի շրջանները փոխանակվում են հոմոլոգ քրոմոսոմների միջև[22][23]: Սա ստեղծում է գեների նոր, հնարավոր է շահավետ համակցություններ, որոնք կարող են սերունդներին էվոլյուցիոն առավելություն տալ[24]: Քրոմոսոմային տրամախաչումը հաճախ սկսվում է այն ժամանակ, երբ Spo11 կոչվող սպիտակուցը ԴՆԹ-ում կատարում է թիրախային երկշղթա ճեղքվածք[25]: Այս վայրերը ոչ պատահականորեն տեղակայված են քրոմոսոմների վրա. սովորաբար միջգենային խթանող շրջաններում և գերադասելիորեն գուանինով և ցիտոզինով հարուստ տիրույթներում[26]։ Այս կրկնակի շղթայական ճեղքման վայրերը հաճախ հանդիպում են ռեկոմբինացիոն թեժ կետերում, շրջաններ քրոմոսոմներում, որոնք ունեն մոտ 1000–2000 բազային զույգ երկարություն և ունեն ռեկոմբինացիայի բարձր արագություն։ Նույն քրոմոսոմի երկու գեների միջև ռեկոմբինացիոն թեժ կետի բացակայությունը հաճախ նշանակում է, որ այդ գեները ապագա սերունդներին կժառանգեն հավասար համամասնությամբ: Սա ցույց է տալիս, որ երկու գեների միջև կապն ավելի մեծ է, քան ակնկալվում էր մեյոզի ընթացքում ինքնուրույն տեսակավորված գեներից[27]:
Միտոտիկ ցիկլում հոմոլոգ ռեկոմբինացիայի ժամանակը
[խմբագրել | խմբագրել կոդը]
Կրկնակի շղթաների ճեղքերը կարող են վերականգնվել հոմոլոգ ռեկոմբինացիայի, պոլիմերազային թետա միջնորդավորված վերջի միացման (TMEJ) կամ ոչ հոմոլոգ վերջի միացման (NHEJ) միջոցով[28]։ NHEJ-ը ԴՆԹ-ի վերականգնման մեխանիզմ է, որը, ի տարբերություն հոմոլոգ ռեկոմբինացիայի, չի պահանջում երկար հոմոլոգ հաջորդականություն՝ ուղղորդելու վերականգնումը: Արդյոք հոմոլոգ ռեկոմբինացիա կամ NHEJ օգտագործվում է կրկնակի շղթաների ճեղքվածքները վերականգնելու համար, հիմնականում որոշվում է բջջային ցիկլի փուլով: Հոմոլոգ ռեկոմբինացիան վերականգնում է ԴՆԹ-ն մինչև բջիջը միտոզ մտնելը (M փուլ): Այն տեղի է ունենում ԴՆԹ-ի վերարտադրության ընթացքում և դրանից կարճ ժամանակ անց, բջջային ցիկլի S և G2 փուլերում, երբ քույր քրոմատիդներն ավելի հեշտությամբ հասանելի են[29]: Համեմատած հոմոլոգ քրոմոսոմների հետ, որոնք նման են մեկ այլ քրոմոսոմին, բայց հաճախ ունեն տարբեր ալելներ, քույր քրոմատիդները իդեալական ձևանմուշ են հոմոլոգ ռեկոմբինացիայի համար, քանի որ դրանք տվյալ քրոմոսոմի նույնական պատճենն են: Երբ հոմոլոգ ձևանմուշը հասանելի չէ կամ երբ ձևանմուշը հնարավոր չէ մուտք գործել հոմոլոգ ռեկոմբինացիայի թերության պատճառով, վնասվածքը վերականգնվում է TMEJ-ի միջոցով բջջային ցիկլի S և G2 փուլերում: Ի տարբերություն հոմոլոգ ռեկոմբինացիայի և TMEJ-ի, NHEJ-ը գերակշռում է բջջային ցիկլի G1 փուլում, երբ բջիջը աճում է, բայց դեռ պատրաստ չէ բաժանման: Այն տեղի է ունենում ավելի քիչ հաճախակի G1 փուլից հետո, բայց պահպանում է առնվազն որոշակի ակտիվություն բջջային ցիկլի ընթացքում: Մեխանիզմները, որոնք կարգավորում են հոմոլոգ ռեկոմբինացիան և NHEJ-ը բջջային ցիկլի ընթացքում, տարբեր տեսակների միջև լայնորեն տարբերվում են[30]:
Ցիկլինից կախված կինազները (CDKs), որոնք փոփոխում են այլ սպիտակուցների ակտիվությունը՝ դրանցում ֆոսֆատ խմբեր ավելացնելով (այսինքն՝ ֆոսֆորիլացնելով), էուկարիոտներում հոմոլոգ վերահամակցման կարևոր կարգավորիչներ են[30]: Երբ ԴՆԹ-ի վերարտադրությունը սկսվում է բողբոջող խմորասնկում, ցիկլինից-կախված կինազ Cdc28-ը սկսում է հոմոլոգ ռեկոմբինացիա՝ ֆոսֆորիլացնելով Sae2 սպիտակուցը[31]: Ֆոսֆատի ավելացումով ակտիվանալուց հետո Sae2-ն առաջացնում է մաքուր կտրվածք ԴՆԹ-ի կրկնակի շղթայի վնասվածքի մոտ: Անհասկանալի է, թե այս կտրվածքի համար պատասխանատու էնդոնուկլեազը հենց Sae2-ն է, թե մեկ այլ սպիտակուց՝ Mre11[32]: Սա թույլ է տալիս սպիտակուցային կոմպլեքսին, ներառյալ Mre11-ին, որը հայտնի է որպես MRX կոմպլեքս, կապվելու ԴՆԹ-ի հետ և սկսել մի շարք սպիտակուցային ռեակցիաներ, որոնք նյութը փոխանակում են ԴՆԹ-ի երկու մոլեկուլների միջև[33]:
Քրոմատինի դերը
[խմբագրել | խմբագրել կոդը]Էուկարիոտային ԴՆԹ-ի քրոմատինի փաթեթավորումը խոչընդոտ է ԴՆԹ-ի վրա հիմնված բոլոր գործընթացների համար, որոնք պահանջում են ֆերմենտների հավաքագրում իրենց գործողության վայրերում: Հոմոլոգ ռեկոմբինացիայի ԴՆԹ-ի վերականգնումը թույլ տալու համար քրոմատինը պետք է վերակառուցվի: Էուկարիոտների մոտ ԱԵՖ-ից կախված քրոմատինային վերականգնող կոմպլեքսները և հիստոն-մոդիֆիկացնող ֆերմենտները երկու գերակշռող գործոններ են, որոնք օգտագործվում են վերափոխման այս գործընթացն իրականացնելու համար[34]:
Քրոմատինի թուլացումն արագորեն տեղի է ունենում ԴՆԹ-ի վնասման վայրում[35]: Ամենավաղ քայլերից մեկում սթրես-ակտիվացված սպիտակուցի կինազը, c-Jun N-տերմինալ կինազը (JNK), ֆոսֆորիլացնում է SIRT6-ը սերին 10-ի վրա՝ ի պատասխան կրկնակի շղթայի ճեղքերի կամ ԴՆԹ-ի այլ վնասների[36]: Հետտրանսլյացիոն այս փոփոխությունը հեշտացնում է SIRT6-ի մոբիլիզացումը ԴՆԹ-ի վնասման վայրերում և պահանջվում է պոլի (ԱԿՖ-ռիբոզ) պոլիմերազ 1-ի (PARP1) արդյունավետ հավաքագրման համար ԴՆԹ-ի կոտրման վայրերում և երկշղթայի վնասվածքների արդյունավետ վերականգնման համար[36]: PARP1 սպիտակուցը սկսում է հայտնվել ԴՆԹ-ի վնասման վայրերում մեկ վայրկյանից պակաս ժամանակում, որի առավելագույն կեսը կուտակվում է վնասի առաջացումից հետո 1,6 վայրկյանի ընթացքում[37]: Այնուհետև Alc1 քրոմատինի վերափոխիչը արագորեն կցվում է PARP1 գործողության արդյունքին, պոլի-ԱԿՖ-ի ռիբոզային շղթային, և Alc1-ն ավարտում է ԴՆԹ-ի վնասը վնասվածքի առաջանալուց 10 վայրկյանի ընթացքում[35]: Քրոմատինի առավելագույն թուլացման մոտ կեսը, ենթադրաբար Alc1-ի գործողության շնորհիվ, տեղի է ունենում 10 վայրկյանում[35]: Այնուհետև դա թույլ է տալիս հավաքագրել ԴՆԹ-ի վերականգնող ֆերմենտը MRE11, որպեսզի սկսի ԴՆԹ վերականգնումը 13 վայրկյանի ընթացքում[37]։
γH2AX, H2AX-ի ֆոսֆորիլացված ձևը նույնպես ներգրավված է ԴՆԹ-ի կրկնակի շղթայի ճեղքումից հետո քրոմատինի դեկոնդենսացմանը տանող վաղ փուլերում: H2AX հիստոնային տարբերակը կազմում է մարդկային քրոմատինի H2A հիստոնների մոտ 10%-ը[38]։ γH2AX (H2AX ֆոսֆորիլացված սերին 139-ի վրա) կարելի է հայտնաբերել բջիջների ճառագայթումից 20 վայրկյան հետո (ԴՆԹ-ի կրկնակի շղթայի ճեղքումով), իսկ γH2AX-ի կես առավելագույն կուտակումը տեղի է ունենում մեկ րոպեում[38]: Ֆոսֆորիլացված γH2AX-ով քրոմատինի ծավալը կազմում է մոտ երկու միլիոն բազային զույգ ԴՆԹ-ի կրկնակի շղթայի ճեղքման վայրում[38]: γH2AX-ն ինքնին չի առաջացնում քրոմատինի խտացում, սակայն ճառագայթումից հետո 30 վայրկյանի ընթացքում RNF8 սպիտակուցը կարող է հայտնաբերվել γH2AX-ի հետ կապված[39]: RNF8-ը միջնորդում է լայնածավալ քրոմատինի դեկոնդենսացիա՝ CHD4-ի հետ իր հետագա փոխազդեցության միջոցով[40], որը նուկլեոսոմի վերափոխման և դեացետիլազային կոմպլեքսի NuRD բաղադրիչն է:
ԴՆԹ-ի վնասման հետևանքով հանգստացում անցնելուց հետո, որին հաջորդում է ԴՆԹ-ի վերականգնումը, քրոմատինը մոտ 20 րոպե հետո վերականգնվում է դեպի խտացման վիճակ, որը մոտ է իր նախավնասման մակարդակին[35]:
Հոմոլոգ ռեկոմբինացիան մեյոզում
[խմբագրել | խմբագրել կոդը]Ողնաշարավորների մոտ այն վայրերը, որտեղ տեղի է ունենում ռեկոմբինացիան, որոշվում են PRDM9-ի միացման վայրերով, մի սպիտակուց, որը ճանաչում է որոշակի հաջորդականության մոտիվը իր ցինկի մատների զանգվածով[41]: Այս տեղամասերում մեկ այլ սպիտակուց՝ SPO11-ը, կատալիզացնում է կրկնակի շղթայի վնասվածքները (DSBs), որոնց մի մասը վերականգնվում է հոմոլոգ քրոմոսոմի հետ վերահամակցման միջոցով: PRDM9-ը կուտակում է և՛ H3K4me3, և՛ H3K36me3 հիստոնային մեթիլացման նշանները այն վայրերում, որտեղ կապվում է, և մեթիլտրանսֆերազային այս ակտիվությունը էական է կրկնակի շղթայի վնասվածքների դիրքավորման մեջ նրա դերի համար: Դրանց ձևավորումից հետո կրկնակի շղթայի վնասված տեղամասերը վերամշակվում են ռեզեկցիայով, ինչի արդյունքում առաջանում է միաշղթա ԴՆԹ (ssDNA), որը զարդարված է DMC1-ով: Միջին զիգոտենից մինչև վաղ պաչիտեն, որպես ռեկոմբինացիոն վերականգնման գործընթացի մաս, DMC1-ը անջատվում է ssDNA-ից և քանակները նվազում են այնքան ժամանակ, մինչև բոլոր կոտրվածքները (բացառությամբ XY քրոմոսոմների) վերականգնվեն ուշ պախիտենի ժամանակ: Այս գործընթացում ներգրավված են մի քանի այլ սպիտակուցներ, այդ թվում՝ ZCWPW1, առաջին սպիտակուցը[42], որն ուղղակիորեն տեղակայված է PRDM9-ի երկակի հիստոնային նշաններով։ ZCWPW1-ը կարևոր է հոմոլոգ կրկնակի շղթայի վնասվածքների վերանորոգման, այլ ոչ թե դիրքավորման համար:
Մոդելներ
[խմբագրել | խմբագրել կոդը]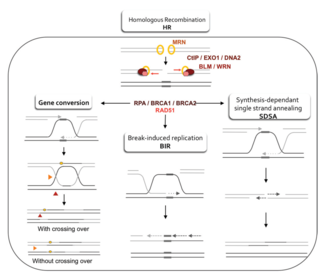
Երկու հիմնական մոդելները, թե ինչպես է հոմոլոգ ռեկոմբինացիան վերականգնում կրկնակի շղթայի ճեղքերը ԴՆԹ-ում, դրանք են կրկնակի շղթայի ճեղքման վերանորոգման ուղին (DSBR) (երբեմն կոչվում է կրկնակի Հոլիդեյի հանգույցի մոդել) և սինթեզից կախված շղթայի ձուլման (SDSA) ուղին[43]: Երկու ուղիները նման են իրենց առաջին մի քանի քայլերում: Կրկնակի շղթայի ճեղքումից հետո MRX կոմպլեքս (մարդկանց MRN համալիրը) կապվում է ԴՆԹ-ի հետ ընդմիջման երկու կողմերում: Այնուհետև կատարվում է ռեզեկցիա, որի ժամանակ կտրվածքի 5' ծայրերի շուրջ ԴՆԹ-ն կտրվում է: Դա տեղի է ունենում երկու հստակ քայլերով. նախ MRX կոմպլեքսը հավաքագրում է Sae2 սպիտակուցը, և այս երկու սպիտակուցները կտրում են 5' ծայրերը ընդմիջման երկու կողմերում՝ ստեղծելով միաշղթա ԴՆԹ-ի կարճ 3' ելուստներ; Երկրորդ քայլում 5'→3' ռեզեկցիան շարունակվում է Sgs1 հելիկազով և Exo1 և Dna2 նուկլեազներով: Որպես հելիկազ, Sgs1-ը «բացում է» երկշղթա ԴՆԹ-ն, մինչդեռ Exo1-ի և Dna2-ի նուկլեազային ակտիվությունը թույլ է տալիս կտրել Sgs1-ի կողմից արտադրված միաշղթա ԴՆԹ-ն[31]։
RPA սպիտակուցը, որն ունի միաշղթա ԴՆԹ-ի հետ բարձր կապակցում, այնուհետև կապում է 3' ելուստները[44]: Մի քանի այլ սպիտակուցների օգնությամբ, որոնք միջնորդում են գործընթացը, Rad51 սպիտակուցը (և Dmc1՝ մեյոզի դեպքում) այնուհետև ձևավորում է նուկլեինասպիտակուցի թելը՝ RPA-ով պատված ԴՆԹ-ի մեկ շղթայի վրա: Այս նուկլեինասպիտակուցային թելիկն այնուհետև սկսում է ԴՆԹ-ի հաջորդականությունների որոնումը, որը նման է 3' վերելքի: Նման հաջորդականություն գտնելուց հետո միաշղթա նուկլեինասպիտակուցային թելիկը տեղափոխվում է (ներխուժում) համանման կամ նույնական ստացողի ԴՆԹ դուպլեքս՝ շղթայի ներխուժում կոչվող գործընթացով: Միտոզով բաժանվող բջիջներում ստացող ԴՆԹ-ի դուպլեքսը սովորաբար քույր քրոմատիդ է, որը նույնական է վնասված ԴՆԹ-ի մոլեկուլին և ապահովում է վերականգնման ձևանմուշ: Մեյոզում, այնուամենայնիվ, ստացող ԴՆԹ-ն հակված է լինել նմանատիպ, բայց ոչ պարտադիր նույնական հոմոլոգ քրոմոսոմից[43]: Շղթայի ներխուժման ժամանակ ձևավորվում է տեղաշարժի հանգույց (D-loop) ներխուժող 3' վերելքի շղթայի և հոմոլոգ քրոմոսոմի միջև: Շղթայի ներխուժումից հետո ԴՆԹ պոլիմերազը երկարացնում է ներխուժող 3' շղթայի վերջը` սինթեզելով նոր ԴՆԹ: Սա փոխում է D հանգույցը խաչաձեւ կառուցվածքի, որը հայտնի է որպես Հոլիդեյյան հանգույց: Դրանից հետո ԴՆԹ-ի ավելի շատ սինթեզ է տեղի ունենում ներխուժող շղթայի վրա (այսինքն՝ սկզբնական 3' ելուստներից մեկը), որն արդյունավետ կերպով վերականգնում է շարանը հոմոլոգ քրոմոսոմի վրա, որը տեղահանվել է շղթայի ներխուժման ժամանակ[43]:
DSBR ուղի
[խմբագրել | խմբագրել կոդը]
Ռեզեկցիայի, շղթայի ներխուժման և ԴՆԹ-ի սինթեզի փուլերից հետո DSBR և SDSA ուղիները դառնում են հստակ[43]: DSBR ուղին եզակի է նրանով, որ երկրորդ 3' ելուստը (որը ներգրավված չէր շղթայի ներխուժման մեջ) նաև կազմում է Հոլիդեյի միացում հոմոլոգ քրոմոսոմի հետ: Կրկնակի Հոլիդեյի հանգույցներն այնուհետև վերածվում են ռեկոմբինացիոն արտադրանքների՝ էնդոնուկլեազների մակարդման միջոցով, սահմանափակող էնդոնուկլեազների մի տեսակ, որը կտրում է միայն մեկ ԴՆԹ շղթա: DSBR ուղին սովորաբար հանգեցնում է տրամախաչման, թեև երբեմն այն կարող է հանգեցնել ոչ տրամախաչված արտադրանքի. վնասված ԴՆԹ-ի մոլեկուլի՝ բաժանված դոնորական տեղանքներից հաջորդականություններ հավաքելու ունակությունը ցուցադրվել է միտոտիկ բողբոջող խմորասնկի դեպքում՝ օգտագործելով պլազմիդներ կամ քրոմոսոմային իրադարձությունների էնդոնուկլեազային ինդուկցիա[45][46]։ Քրոմոսոմային տրամախաչման այս հակման պատճառով DSBR ուղին հավանական մոդել է այն բանի, թե ինչպես է տեղի ունենում խաչաձև հոմոլոգ ռեկոմբինացիա մեյոզի ժամանակ[22]:
Արդյո՞ք DSBR ուղու մեջ ռեկոմբինացումը հանգեցնում է քրոմոսոմային տրամախաչման, որոշվում է նրանով, թե ինչպես է կրկնակի Հոլիդեյ հանգույցը կտրվում կամ «լուծվում»: Քրոմոսոմային տրամախաչում տեղի կունենա, եթե Հոլիդեյի մի հանգույցը կտրվի հատվող շղթայի վրա, իսկ մյուս Հոլիդեյ հանգույցը կտրվի չհատվող շղթայի վրա (Նկար 5-ում հորիզոնական մանուշակագույն սլաքների երկայնքով Հոլիդեյի մի հանգույցում և ուղղահայաց նարնջագույն սլաքների երկայնքով՝ մյուսում: ) Այլապես, եթե Հոլիդեյի երկու հանգույցները կտրված են հատվող թելերի վրա (Նկար 5-ում նշված երկու Հոլիդեյի հանգույցներում հորիզոնական մանուշակագույն սլաքների երկայնքով), ապա առանց տրամախաչման քրոմոսոմներ կստեղծվեն[47]:
SDSA ուղի
[խմբագրել | խմբագրել կոդը]Հոմոլոգ ռեկոմբինացիա SDSA ուղու միջոցով տեղի է ունենում բջիջներում, որոնք բաժանվում են միտոզի և մեյոզի միջոցով և հանգեցնում ոչ տրամախաչված արտադրանքի: Այս մոդելում ներխուժող 3' շղթան երկարացվում է ստացող ԴՆԹ-ի երկայնքով ԴՆԹ պոլիմերազի միջոցով և ազատվում է, երբ դոնորի և ստացողի ԴՆԹ-ի մոլեկուլների Հոլիդեյի հանգույցը սահում է մի գործընթացում, որը կոչվում է ճյուղային միգրացիա: Ներխուժող շղթայի նոր սինթեզված 3' ծայրն այնուհետև կարող է կցել վնասված քրոմոսոմի մյուս 3' ծայրամասին՝ լրացուցիչ հիմքերի զուգավորման միջոցով: Շղթաների ձուլումից հետո երբեմն կարող է մնալ ԴՆԹ-ի մի փոքր ծալք: Ցանկացած նման փեղկեր հանվում են, և SDSA-ի ուղին ավարտվում է մնացած միաշղթա բացերի վերակնքումով, որը նաև հայտնի է որպես կապակցում[48]:
Միտոզի ընթացքում ԴՆԹ-ի կրկնակի շղթայական ճեղքվածքները վերականգնելու հիմնական հոմոլոգ ռեկոմբինացիոն ուղին, ըստ երևույթին, SDSA ուղին է (այլ ոչ թե DSBR ուղին)[49]: SDSA ուղին արտադրում է ոչ տրամախաչված ռեկոմբինանտներ (Նկար 5): Մեյոզի ժամանակ հաճախակի են լինում նաև ոչ տրամախաչված ռեկոմբինանտներ, որոնք, ըստ երևույթին, առաջանում են հիմնականում նաև SDSA ուղու միջոցով[49][50]: Ոչ տրամախաչված ռեկոմբինացիոն իրադարձությունները, որոնք տեղի են ունենում մեյոզի ընթացքում, հավանաբար արտացոլում են ԴՆԹ-ի կրկնակի շղթայի վնասների կամ ԴՆԹ-ի այլ տեսակի վնասների վերականգնման դեպքերը:
SSA ուղի
[խմբագրել | խմբագրել կոդը]
Հոմոլոգ ռեկոմբինացիայի միաշղթա ձուլման (SSA) ուղին վերականգնում է կրկնակի շղթայի ընդմիջումները երկու կրկնվող հաջորդականությունների միջև: SSA ուղին եզակի է նրանով, որ այն չի պահանջում ԴՆԹ-ի առանձին նմանատիպ կամ նույնական մոլեկուլ, ինչպես DSBR կամ SDSA հոմոլոգ ռեկոմբինացիայի ուղիները: Փոխարենը, SSA ուղին պահանջում է միայն մեկ ԴՆԹ դուպլեքս և օգտագործում է կրկնվող հաջորդականությունները որպես նույնական հաջորդականություններ, որոնք հոմոլոգ ռեկոմբինացիային անհրաժեշտ են վերանորոգման համար: Ճանապարհը համեմատաբար պարզ է հայեցակարգով. այն բանից հետո, երբ միևնույն ԴՆԹ-ի երկու շղթաները կտրվում են կրկնակի շղթայի ճեղքման վայրի շուրջ, արդյունքում ստացված երկու 3' ելուստները, այնուհետև հարթվում և կռվում են միմյանց հետ՝ վերականգնելով ԴՆԹ-ն որպես շարունակական դուպլեքս[48][51]:
Քանի որ ԴՆԹ-ն կրկնակի շղթայի ճեղքվածքի շուրջը կտրվում է, արտադրվող միաշղթա 3' ելուստները պատված են RPA սպիտակուցով, ինչը թույլ չի տալիս 3' ելուստներին կպչել իրար վրա[52]: Այնուհետև Rad52 կոչվող սպիտակուցը կապում է կրկնվող հաջորդականություններից յուրաքանչյուրը ընդմիջման երկու կողմերում և հարթեցնում է դրանք, որպեսզի երկու լրացուցիչ կրկնվող հաջորդականությունները կարողանան ձուլվել[52]: Ձուլման ավարտից հետո 3' ելուստների մնացած ոչ հոմոլոգ փեղկերը կտրվում են մի շարք նուկլեազների միջոցով, որոնք հայտնի են որպես Rad1/Rad10, որոնք բերվում են փեղկերին Saw1 և Slx4 սպիտակուցների միջոցով[52][53]: ԴՆԹ-ի նոր սինթեզը լրացնում է ցանկացած բաց, և կապակցումը վերականգնում է ԴՆԹ-ի դուպլեքսը որպես երկու շարունակական շղթա[54]: ԴՆԹ-ի հաջորդականությունը կրկնությունների միջև միշտ կորչում է, ինչպես և երկու կրկնություններից մեկը: SSA ուղին համարվում է մուտագեն, քանի որ այն հանգեցնում է գենետիկական նյութի նման ջնջումների[48]:
BIR ուղի
[խմբագրել | խմբագրել կոդը]ԴՆԹ-ի կրկնապատկման ժամանակ կրկնապատկման պատառաքաղներում երբեմն կարող են հանդիպել կրկնակի շղթաների ընդմիջումներ, քանի որ ԴՆԹ հելիկազը բացում է կաղապարի շարանը: Այս թերությունները վերականգնվում են հոմոլոգ ռեկոմբինացիայի վնասվածքից առաջացած վերարտադրության (BIR) ճանապարհով: BIR ճանապարհի ճշգրիտ մոլեկուլային մեխանիզմները մնում են անհասկանալի: Առաջարկվող երեք մեխանիզմները որպես սկզբնական քայլ ունեն շղթայի ներխուժումը, սակայն դրանք տարբերվում են նրանով, թե ինչպես են մոդելավորում D-հանգույցի միգրացիան և վերահամակցման հետագա փուլերը[55]:
BIR ուղին կարող է նաև օգնել պահպանել թելոմերների երկարությունը (ԴՆԹ-ի շրջանները էուկարիոտիկ քրոմոսոմների վերջում) թելոմերազի բացակայության դեպքում (կամ հետ համագործակցելով): Առանց թելոմերազ ֆերմենտի գործող պատճենների, թելոմերները սովորաբար կրճատվում են միտոզի յուրաքանչյուր ցիկլով, որն ի վերջո արգելափակում է բջիջների բաժանումը և հանգեցնում ծերացման: Բողբոջող խմորասնկի բջիջներում, որտեղ թելոմերազն ապաակտիվացվել է մուտացիաների միջոցով, նկատվել են երկու տեսակի «վերապրած» բջիջներ՝ կանխելու սպասվածից երկար ծերացումը՝ երկարացնելով իրենց թելոմերները BIR ուղիներով[55]:
Թելոմերների երկարության պահպանումը չափազանց կարևոր է բջիջների անմահացման համար՝ քաղցկեղի հիմնական հատկանիշը: Քաղցկեղների մեծ մասը պահպանում է թելոմերները՝ վերկարգավորելով թելոմերազը: Այնուամենայնիվ, մարդու քաղցկեղի մի քանի տեսակների դեպքում BIR-ի նման ուղին օգնում է պահպանել որոշ ուռուցքներ՝ գործելով որպես թելոմերների պահպանման այլընտրանքային մեխանիզմ[56]: Այս փաստը գիտնականներին ստիպել է հետաքննել, թե արդյոք թելոմերների պահպանման նման ռեկոմբինացիայի վրա հիմնված մեխանիզմները կարող են խափանել հակաքաղցկեղային դեղամիջոցները, ինչպիսիք են թելոմերազի ինհիբիտորները[57]:
Բակտերիաներում
[խմբագրել | խմբագրել կոդը]
Հոմոլոգ ռեկոմբինացիան բակտերիաների ԴՆԹ-ի վերականգնման հիմնական գործընթացն է: Այն նաև կարևոր է բակտերիաների պոպուլյացիաներում գենետիկական բազմազանություն առաջացնելու համար, թեև գործընթացը էականորեն տարբերվում է մեյոտիկ ռեկոմբինացիայից, որը վերականգնում է ԴՆԹ-ի վնասները և բազմազանություն է բերում էուկարիոտների գենոմներում: Հոմոլոգ ռեկոմբինացիան ամենաշատն ուսումնասիրվել է և լավագույնս հասկանալի է Escherichia coli-ի համար[59]: Բակտերիաներում ԴՆԹ-ի երկշղթա կոտրվածքները վերականգնվում են հոմոլոգ ռեկոմբինացիայի RecBCD ճանապարհով: Ենթադրվում է, որ վնասվածքները, որոնք տեղի են ունենում ԴՆԹ-ի երկու շղթաներից միայն մեկի վրա, որոնք հայտնի են որպես մեկ շղթայական բացեր, վերականգնվում են RecF ճանապարհով[60]: Ե՛վ RecBCD, և՛ RecF ուղիները ներառում են մի շարք ռեակցիաներ, որոնք հայտնի են որպես ճյուղային միգրացիա, որտեղ ԴՆԹ-ի առանձին շղթաները փոխանակվում են դուպլեքս ԴՆԹ-ի երկու խաչաձև մոլեկուլների միջև և լուծվում են, որոնցում ԴՆԹ-ի այդ երկու խաչաձև մոլեկուլները կտրվում են և վերականգնվում իրենց նորմալ երկշղթա վիճակին:
RecBCD ուղի
[խմբագրել | խմբագրել կոդը]

RecBCD ուղին հիմնական ռեկոմբինացիոն ուղին է, որն օգտագործվում է շատ բակտերիաների մեջ՝ վերականգնելու ԴՆԹ-ի կրկնակի շղթայի ճեղքերը, և սպիտակուցները հայտնաբերված են բակտերիաների լայն տեսականիում[63][64][65]: Այս կրկնակի շղթայի ճեղքերը կարող են առաջանալ ուլտրամանուշակագույն լույսի և այլ ճառագայթման, ինչպես նաև քիմիական մուտագենների պատճառով: Կրկնակի շղթաների ճեղքեր կարող են առաջանալ նաև ԴՆԹ-ի վերարտադրման արդյունքում՝ մեկ շղթայական անցքի կամ բացվածքի միջոցով: Նման իրավիճակն առաջացնում է այն, ինչը հայտնի է որպես փլուզված վերարտադրության պատառաքաղ և ամրագրվում է հոմոլոգ ռեկոմբինացիայի մի քանի ուղիներով, ներառյալ RecBCD ուղին[66]:
Այս ճանապարհով, RecBCD կոչվող երեք ենթամիավոր ֆերմենտային կոմպլեքսը սկսում է ռեկոմբինացիա՝ կապվելով երկշղթա ԴՆԹ-ի բեկման բութ կամ գրեթե բութ ծայրին: Այն բանից հետո, երբ RecBCD-ն կապում է ԴՆԹ-ի ծայրը, RecB և RecD ենթամիավորները սկսում են անջատել ԴՆԹ-ի դուպլեքսը հելիկազի ակտիվության միջոցով: RecB ենթամիավորն ունի նաև նուկլեազային տիրույթ, որը կտրում է ԴՆԹ-ի միայնակ շղթան, որը առաջանում է անջատման գործընթացից: Այս անջատումը շարունակվում է այնքան ժամանակ, մինչև RecBCD-ն չհանդիպի հատուկ նուկլեոտիդային հաջորդականության (5'-GCTGGTGG-3'), որը հայտնի է որպես Չի-ի տեղամաս[65]:
Չի տեղամասի հանդիպելիս RecBCD ֆերմենտի ակտիվությունը կտրուկ փոխվում է[64][61][67]։ ԴՆԹ-ի լուծարումը դադարում է մի քանի վայրկյանով և այնուհետև վերսկսվում սկզբնական արագության մոտավորապես կեսով: Հավանաբար, դա տեղի է ունենում այն պատճառով, որ ավելի դանդաղ RecB հելիկազն արձակում է ԴՆԹ-ն Չի-ից հետո, այլ ոչ թե ավելի արագ RecD հելիկազն, որը քանդում է ԴՆԹ-ն Չի-ից առաջ[68][69]: Չի-ի տեղամասի ճանաչումը նաև փոխում է RecBCD ֆերմենտը այնպես, որ այն կտրում է ԴՆԹ-ի շարանը Չի-ով և սկսում է բեռնել բազմաթիվ RecA սպիտակուցներ միաշղթա ԴՆԹ-ի վրա նոր առաջացած 3' ծայրով: Ստացված RecA-ով պատված նուկլեոսպիտակուցի թելիկն այնուհետև որոնում է ԴՆԹ-ի նմանատիպ հաջորդականությունները հոմոլոգ քրոմոսոմի վրա: Որոնման գործընթացը հրահրում է ԴՆԹ-ի դուպլեքսի ձգումը, որն ուժեղացնում է հոմոլոգիայի ճանաչումը (մեխանիզմ, որը կոչվում է կոնֆորմացիոն սրբագրում)[70][71][72]: Նման հաջորդականություն գտնելով՝ միաշղթա նուկլեոսպիտակուցային թելիկը շարժվում է դեպի հոմոլոգ ստացող ԴՆԹ-ի դուպլեքս՝ մի գործընթացով, որը կոչվում է շղթաների ներխուժում[73]: Ներխուժող 3' վերելքը հանգեցնում է նրան, որ ստացող ԴՆԹ-ի դուպլեքսի շղթաներից մեկը տեղաշարժվում է և ձևավորվում է D հանգույց: Եթե D-հանգույցը կտրված է, թելերի մեկ այլ փոխանակում ձևավորում է խաչաձև կառուցվածք, որը կոչվում է Հոլիդեյյան հանգույց[65]: Հոլիդեյի հանգույցի լուծումը RuvABC-ի կամ RecG-ի ինչ-որ համակցության միջոցով կարող է առաջացնել երկու ռեկոմբինանտ ԴՆԹ մոլեկուլ՝ փոխադարձ գենետիկ տիպերով, եթե փոխազդող ԴՆԹ-ի երկու մոլեկուլները գենետիկորեն տարբերվում են: Որպես այլընտրանք, Չիի մոտ ներխուժող 3' ծայրը կարող է առաջացնել ԴՆԹ սինթեզ և ձևավորել կրկնօրինակման պատառաքաղ: Այս տեսակի լուծումը արտադրում է միայն մեկ տեսակի ռեկոմբինանտ (ոչ փոխադարձ):
RecF ուղի
[խմբագրել | խմբագրել կոդը]Բակտերիաները, ըստ երևույթին, օգտագործում են հոմոլոգ ռեկոմբինացիայի RecF ուղին ԴՆԹ-ում միաշղթա բացերը վերականգնելու համար: Երբ RecBCD ուղին ապաակտիվացվում է մուտացիաներով, իսկ լրացուցիչ մուտացիաներն անակտիվացնում են SbcCD և ExoI նուկլեազները, RecF ուղին կարող է նաև վերականգնել ԴՆԹ-ի երկշղթայի ճեղքերը[74]: RecF ճանապարհում RecQ հելիկազն արձակում է ԴՆԹ-ն, իսկ RecJ նուկլեազը քայքայում է շարանը 5' ծայրով, թողնելով շարանը 3' ծայրով անձեռնմխելի: RecA սպիտակուցը կապվում է այս շղթայի հետ և կամ օգնում է RecF, RecO և RecR սպիտակուցներին, կամ կայունանում է դրանցով: RecA նուկլեոսպիտակուցի թելիկն այնուհետև փնտրում է հոմոլոգ ԴՆԹ և փոխանակում տեղերը նույնական կամ գրեթե նույնական շղթայի հետ հոմոլոգ ԴՆԹ-ում:
Թեև դրանց սկզբնական փուլերում ներգրավված սպիտակուցները և հատուկ մեխանիզմները տարբեր են, երկու ուղիները նման են նրանով, որ երկուսն էլ պահանջում են միաշղթա ԴՆԹ 3' ծայրով և RecA սպիտակուցը շղթայի ներխուժման համար: Երթուղիները նման են նաև ճյուղային միգրացիայի իրենց փուլերին, որոնցում Հոլիդեյի հանգույցը սահում է մեկ ուղղությամբ, և վերկանգնմամբ, որտեղ Հոլիդեյի հանգույցները բաժանվում են ֆերմենտների միջոցով[75][76]: Վերկանգնմաան այլընտրանքային, ոչ փոխադարձ տեսակը կարող է առաջանալ նաև ցանկացած ճանապարհով:
Ճյուղային միգրացիա
[խմբագրել | խմբագրել կոդը]Շղթայի ներխուժումից անմիջապես հետո Հոլիդեյի հանգույցը շարժվում է կապված ԴՆԹ-ի երկայնքով ճյուղային միգրացիայի գործընթացում: Հոլիդեյի հանգույցի այս շարժման մեջ է, որ հիմքերի զույգերը փոխանակվում են երկու հոմոլոգ ԴՆԹ-ի դուպլեքսների միջև։ Ճյուղային միգրացիան կատալիզացնելու համար RuvA սպիտակուցը նախ ճանաչում և կապվում է Հոլիդեյ հանգույցին և հավաքագրում RuvB սպիտակուցը՝ ձևավորելու RuvAB համալիրը: RuvB սպիտակուցի երկու խումբ, որոնցից յուրաքանչյուրը կազմում է օղակաձեւ Ադենոզին եռֆոսֆատազ, բեռնված են Հոլիդեյի հանգույցի հակառակ կողմերում, որտեղ նրանք գործում են որպես զույգ պոմպեր, որոնք ապահովում են ճյուղերի միգրացիայի ուժը: RuvB-ի այդ երկու օղակների միջև RuvA սպիտակուցի երկու խումբ հավաքվում է Հոլիդեյ հանգույցի կենտրոնում այնպես, որ հանգույցում գտնվող ԴՆԹ-ն խցկված է RuvA-ի յուրաքանչյուր խմբի միջև: ԴՆԹ-ի երկու դուպլեքսների շղթաները՝ «դոնոր» և «ստացող» դուպլեքսները, բացվում են RuvA-ի մակերեսի վրա, քանի որ դրանք ուղղորդվում են սպիտակուցի կողմից մի դուպլեքսից մյուսը[77][78]:
Վերականգնում
[խմբագրել | խմբագրել կոդը]Ռեկոմբինացիայի վերակնգնման փուլում, շղթայի ներխուժման գործընթացի արդյունքում ձևավորված Հոլիդեյի ցանկացած հանգույց կտրվում է, դրանով իսկ վերականգնելով երկու առանձին ԴՆԹ մոլեկուլներ: Այս ճեղքումը կատարվում է RuvAB համալիրի միջոցով, որը փոխազդում է RuvC-ի հետ, որոնք միասին կազմում են RuvABC համալիրը: RuvC-ն էնդոնուկլեազ է, որը կտրում է դեգեներատիվ հաջորդականությունը 5'-(A/T)TT(G/C)-3': Հերթականությունը հաճախ հանդիպում է ԴՆԹ-ում, մոտավորապես 64 նուկլեոտիդից մեկ անգամ[78]: Նախքան կտրելը, RuvC-ն, ամենայն հավանականությամբ, մուտք է ստանում դեպի Հոլիդեյ հանգույց՝ տեղաշարժելով այնտեղ գտնվող երկու RuvA տետրամերներից մեկը, որը ծածկում է ԴՆԹ-ն[77]։ Ռեկոմբինացիան հանգեցնում է կամ «կտրված» կամ «կարկատված» արդյունքների՝ կախված նրանից, թե RuvC-ն ինչպես է կտրում Հոլիդեյ հանգույցը[78]: Կտրված արդյունքները տրամախաչման արդյունք են, որոնցում տեղի է ունենում գենետիկական նյութի վերադասավորում ռեկոմբինացման վայրի շուրջ: Մյուս կողմից, կարկատված արդյունքները չտրամախաչված արտադրանք են, որոնցում նման վերադասավորում չկա, և կա միայն հիբրիդային ԴՆԹ-ի «կարկատան» ռեկոմբինացիոն արտադրանքում[79]:
Հեշտացնելով գենետիկական փոխանցումը
[խմբագրել | խմբագրել կոդը]Հոմոլոգ ռեկոմբինացիան դոնորային ԴՆԹ-ի ինտեգրման կարևոր մեթոդ է ստացող օրգանիզմի գենոմում հորիզոնական գեների փոխանցման գործընթացում, որի միջոցով օրգանիզմը ներառում է օտար ԴՆԹ-ն այլ օրգանիզմից՝ առանց այդ օրգանիզմի սերունդ լինելու: Հոմոլոգ ռեկոմբինացիան պահանջում է, որ մուտքային ԴՆԹ-ն շատ նման լինի ստացողի գենոմին, և այդ պատճառով գեների հորիզոնական փոխանցումը սովորաբար սահմանափակվում է նմանատիպ բակտերիաներով[80]: Բակտերիաների մի քանի տեսակների ուսումնասիրությունները ցույց են տվել, որ կա ռեկոմբինացիայի հաճախականության լոգարիթմական-գծային նվազում՝ հյուրընկալող և ստացող ԴՆԹ-ի միջև հաջորդականության աճով[81][82][83]:
Բակտերիաների կոնյուգացիայի դեպքում, որտեղ ԴՆԹ-ն փոխանցվում է բակտերիաների միջև բջիջ-բջիջ անմիջական շփման միջոցով, հոմոլոգ ռեկոմբինացիան օգնում է օտար ԴՆԹ-ի ինտեգրմանը հյուրընկալող գենոմի մեջ RecBCD ճանապարհի միջոցով: RecBCD ֆերմենտը նպաստում է ռեկոմբինացիային այն բանից հետո, երբ ԴՆԹ-ն վերածվում է միաշղթա ԴՆԹ-ից, որի ձևով այն սկզբնապես մտնում է բակտերիա, կրկնօրինակման ընթացքում կրկնակի շղթայական ԴՆԹ-ի: RecBCD ուղին կարևոր է նաև փոխակերպման վերջնական փուլի համար՝ հորիզոնական գեների փոխանցման մի տեսակ, որի ժամանակ ԴՆԹ-ն վիրուսի միջոցով փոխանցվում է մի բակտերիայից մյուսը: Օտար, բակտերիալ ԴՆԹ-ն երբեմն սխալ կերպով ներկառուցվում է բակտերիոֆագ վիրուսի կապսիդների գլխի մեջ, քանի որ ԴՆԹ-ն փաթեթավորվում է նոր բակտերիոֆագների մեջ վիրուսային վերարտադրության ժամանակ: Երբ այս նոր բակտերիոֆագները վարակում են այլ բակտերիաներ, նախորդ հյուրընկալ բակտերիայից ԴՆԹ-ն ներարկվում է նոր բակտերիային հյուրընկալողին՝ որպես երկշղթա ԴՆԹ: RecBCD ֆերմենտը այնուհետև ներառում է այս երկշղթա ԴՆԹ-ն նոր բակտերիալ հյուրընկալողի գենոմի մեջ[65]:
Բակտերիալ փոխակերպում
[խմբագրել | խմբագրել կոդը]Բնական բակտերիաների փոխակերպումը ներառում է ԴՆԹ-ի փոխանցումը դոնոր բակտերիայից դեպի ստացող բակտերիա, որտեղ և՛ դոնորը, և՛ ստացողը սովորաբար նույն տեսակից են: Փոխակերպումը, ի տարբերություն բակտերիաների կոնյուգացիայի և տրանսդուկցիայի, կախված է բազմաթիվ բակտերիաների գենային արտադրանքներից, որոնք հատուկ փոխազդում են այս գործընթացն իրականացնելու համար[84]: Այսպիսով, փոխակերպումը ակնհայտորեն բակտերիալ հարմարեցում է ԴՆԹ-ի փոխանցման համար: Որպեսզի բակտերիան միանա, ընդունի և ինտեգրի դոնորային ԴՆԹ-ն իր ռեզիդենտ քրոմոսոմին հոմոլոգ ռեկոմբինացիայի միջոցով, այն նախ պետք է մտնի հատուկ ֆիզիոլոգիական վիճակ, որը կոչվում է կոմպետենտություն: RecA/Rad51/DMC1 գեների ընտանիքը կենտրոնական դեր է խաղում բակտերիաների փոխակերպման ժամանակ հոմոլոգ ռեկոմբինացիայի մեջ, ինչպես դա անում է էուկարիոտիկ մեյոզի և միտոզի ժամանակ: Օրինակ, RecA սպիտակուցը էական նշանակություն ունի Bacillus subtilis-ում և Streptococcus pneumoniae-ում[85] փոխակերպման համար, և RecA գենի էքսպրեսիան առաջանում է այս օրգանիզմների փոխակերպման կոմպետենտության զարգացման ընթացքում:
Որպես փոխակերպման գործընթացի մաս, RecA սպիտակուցը փոխազդում է միաշղթա ԴՆԹ մուտք գործելու հետ՝ ձևավորելով RecA/միաշղթա ԴՆԹ միջուկներ, որոնք սկանավորում են ռեզիդենտ քրոմոսոմը հոմոլոգիայի շրջանների համար և ներմուծող միաշղթա ԴՆԹ-ն բերում համապատասխան շրջան, որտեղ շղթաների փոխանակում և հոմոլոգ ռեկոմբինացիա է տեղի ունենում[86]։ Այսպիսով, բակտերիաների փոխակերպման ժամանակ հոմոլոգ ռեկոմբինացիայի գործընթացը հիմնարար նմանություններ ունի մեյոզի ժամանակ հոմոլոգ ռեկոմբինացիայի հետ:
Վիրուսներում
[խմբագրել | խմբագրել կոդը]Հոմոլոգ ռեկոմբինացիա տեղի է ունենում վիրուսների մի քանի խմբերում: ԴՆԹ-ի վիրուսներում, ինչպիսին է հերպեսի վիրուսը, ռեկոմբինացիան տեղի է ունենում ճեղքման և վերամիավորման մեխանիզմի միջոցով, ինչպես բակտերիաներում և էուկարիոտներում[87]: Կան նաև ապացույցներ որոշ ՌՆԹ վիրուսների, մասնավորապես դրական զգացողությամբ միաշղթա ՌՆԹ վիրուսների, ինչպիսիք են ռետրովիրուսները, պիկորնավիրուսները և կորոնավիրուսները, ռեկոմբինացիայի վերաբերյալ[88]: Տարաձայնություններ կան այն հարցի շուրջ, թե արդյոք հոմոլոգ ռեկոմբինացիա տեղի է ունենում բացասական զգացողությամբ միաշղթա ՌՆԹ վիրուսներում, ինչպիսին է գրիպը[89]:
ՌՆԹ վիրուսների դեպքում հոմոլոգ ռեկոմբինացիան կարող է լինել կամ ճշգրիտ կամ ոչ ճշգրիտ: ՌՆԹ-ՌՆԹ ռեկոմբինացիայի ճշգրիտ տիպում տարբերություն չկա ծնողական ՌՆԹ-ի երկու հաջորդականությունների և ստացված տրամախաչված ՌՆԹ շրջանի միջև: Դրա պատճառով հաճախ դժվար է որոշել տրամախաչային իրադարձությունների գտնվելու վայրը երկու վերամիավորվող ՌՆԹ հաջորդականությունների միջև: ՌՆԹ-ի ոչ ճշգրիտ հոմոլոգ ռեկոմբինացիայի դեպքում տրամախաչման շրջանը որոշակի տարբերություն ունի ծնողական ՌՆԹ-ի հաջորդականությունների հետ, ինչը պայմանավորված է նուկլեոտիդների ավելացումով, ջնջմամբ կամ այլ փոփոխությամբ: Տրամախաչման ճշգրտության մակարդակը վերահսկվում է ՌՆԹ-ի երկու վերամիավորվող շղթաների հաջորդականության համատեքստով. ադենինով և ուրացիլով հարուստ հաջորդականությունները նվազեցնում են տրամախաչման ճշգրտությունը[88][90]:
Հոմոլոգ ռեկոմբինացիան կարևոր է վիրուսների էվոլյուցիան հեշտացնելու համար[88][91]: Օրինակ, եթե տարբեր անբարենպաստ մուտացիաներով երկու վիրուսների գենոմները ենթարկվում են ռերակոմբինացման, ապա նրանք կարող են վերականգնել լիովին ֆունկցիոնալ գենոմը: Որպես այլընտրանք, եթե երկու նմանատիպ վիրուսներ վարակել են նույն ընդունող բջիջը, ապա հոմոլոգ ռեկոմբինացիան կարող է թույլ տալ այդ երկու վիրուսներին փոխանակել գեները և այդպիսով զարգացնել իրենց ավելի հզորարբերակները[91]:
Հոմոլոգ ռեկոմբինացիան առաջարկված մեխանիզմն է, որի միջոցով ԴՆԹ վիրուսը մարդու հերպեսի վիրուս-6-ն ինտեգրվում է մարդու թելոմերներին[92]:
Երբ երկու կամ ավելի վիրուսներ, որոնցից յուրաքանչյուրը պարունակում է մահացու գենոմային վնաս, վարակում են նույն ընդունող բջիջը, վիրուսի գենոմները հաճախ կարող են զուգակցվել միմյանց հետ և ենթարկվել հոմոլոգ ռեկոմբինացիոն վերանորոգման՝ կենսունակ սերունդ առաջացնելու համար: Այս գործընթացը, որը հայտնի է որպես բազմակի վերաակտիվացում, ուսումնասիրվել է մի քանի բակտերիոֆագերի, այդ թվում՝ T4 ֆագի մոտ[93]: T4 ֆագի դեպքում ռեկոմբինացիոն վերականգնման համար օգտագործվող ֆերմենտները ֆունկցիոնալորեն հոմոլոգ են բակտերիաների և էուկարիոտների ռեկոմբինացիոն վերանորոգման մեջ օգտագործվող ֆերմենտների հետ[94]: Մասնավորապես, ինչ վերաբերում է շղթայի փոխանակման ռեակցիայի համար անհրաժեշտ գենին, որը հոմոլոգ ռեկոմբինացիոն վերականգնման առանցքային քայլ է, կա վիրուսներից մարդկանց ֆունկցիոնալ հոմոլոգիա (այսինքն՝ uvsX ֆագում T4; recA E. coli-ում և այլ բակտերիաներում, և rad51 և dmc1 խմորասնկի և այլ էուկարիոտների, ներառյալ մարդկանց մեջ)[95]։ Բազմապատկության վերաակտիվացումը նույնպես ցուցադրվել է բազմաթիվ պաթոգեն վիրուսների դեպքում[96]:
Կորոնավիրուս
[խմբագրել | խմբագրել կոդը]Կորոնավիրուսները ունակ են գենետիկ ռեկոմբինացիայի, երբ առնվազն երկու վիրուսային գենոմ կա նույն վարակված բջջում։ ՌՆԹ-ի ռեկոմբինացիան, ըստ երևույթին, հիմնական շարժիչ ուժն է (1) CoV տեսակի գենետիկական փոփոխականությունը որոշելու համար, (2) CoV տեսակի մի հյուրընկալողից մյուսը անցնելու կարողությունը և (3) հազվադեպ՝ նոր CoV-ների առաջացումը[97]: CoV-ներում ռեկոմբինացիայի մեխանիզմը, ամենայն հավանականությամբ, ներառում է կաղապարի փոխարկումը գենոմի վերարտադրության ժամանակ[97]: ՌՆԹ վիրուսներում ռեկոմբինացիան, ըստ երևույթին, հարմարեցում է գենոմի վնասը հաղթահարելու համար[98]:
SARS-CoV-2 համաճարակի ընկալիչների հետ կապող ամբողջ մոտիվը, ըստ երևույթին, ներդրվել է պանգոլինների կորոնավիրուսներից ռեկոմբինացիայի միջոցով[99]: Նման ռեկոմբինացիոն իրադարձությունը կարող է կարևոր քայլ լինել SARS-CoV-2-ի՝ մարդկանց վարակելու ունակության էվոլյուցիայում[99]: Ռեկոմբինացիոն իրադարձությունները, հավանաբար, հիմնական քայլերն են էվոլյուցիոն գործընթացում, որը հանգեցնում է նոր մարդկային կորոնավիրուսների առաջացմանը[100]:
2020 թվականին COVID-19 համաճարակի ժամանակ ավստրալիական SARS-CoV-2 մեկուսացման գենոմային շատ հաջորդականություններ ունենում են ջնջումներ կամ մուտացիաներ (29742G>A կամ 29742G>U; «G19A» կամ «G19U») Coronavirus 3' ցողունային հանգույցում II- ինչպես մոտիվը (s2m), ՌՆԹ-ի մոտիվ վիրուսի գենոմի 3' չթարգմանված հատվածում, ինչը ենթադրում է, որ ՌՆԹ-ի ռեկոմբինացիայի իրադարձությունները կարող են տեղի ունենալ SARS-CoV-2-ի s2m-ում: 1319 Ավստրալիայի SARS‐CoV‐2 հաջորդականությունների հաշվարկային վերլուծության հիման վրա՝ օգտագործելով Recco ալգորիթմը (https://recco.bioinf.mpi-inf.mpg.de/ Արխիվացված 2024-03-21 Wayback Machine), 29742G («G19»), 29744G («G21») և 29751G («G28») կանխատեսվել էին որպես ռեկոմբինացիոն թեժ կետեր[101]:
SARS-CoV-2-ի բռնկումը Diamond Princess նավի վրա, ամենայն հավանականությամբ, առաջացել է կամ մեկ անձից, որը վարակվել է վիրուսի տարբերակով, որը նույնական է Ուհանի WIV04 մեկուսացվածներին, կամ միաժամանակ մեկ այլ առաջնային դեպքի հետ, որը վարակված է 11083G > T մուտացիա պարունակող վիրուսով: Կապի անհավասարակշռության վերլուծությունը հաստատեց, որ ՌՆԹ-ի ռեկոմբինացումը 11083G > T մուտացիայի հետ նույնպես նպաստել է վիրուսային սերունդների շրջանում մուտացիաների ավելացմանը: Գտածոները ցույց են տալիս, որ SARS-CoV-2-ի 11083G > T մուտացիան տարածվել է նավի կարանտինի ժամանակ և առաջացել է նոր ՌՆԹ-ի ռեկոմբինացման միջոցով՝ դրական ընտրության ճնշման ներքո: Բացի այդ, այս նավարկության երեք հիվանդների մոտ երկու մուտացիա՝ 29736G > T և 29751G > T («G13» և «G28») նույնպես տեղաբաշխված են եղել Coronavirus 3′ ցողունային հանգույց II-ի նմանվող մոտիվում (s2m), որպես «G28»: կանխատեսվում էր որպես ավստրալական SARS-CoV-2 մուտանտների ռեկոմբինացիոն թեժ կետեր: Չնայած s2m-ը համարվում է ՌՆԹ-ի մոտիվ, որը խիստ պահպանված է բազմաթիվ կորոնավիրուսային տեսակների մեջ, այս արդյունքը նաև ենթադրում է, որ SARS-CoV-2-ի s2m-ը ավելի շուտ ՌՆԹ-ի ռեկոմբինացիայի/մուտացիայի թեժ կետ է[102]:
Դիսֆունկցիայի հետևանքները
[խմբագրել | խմբագրել կոդը]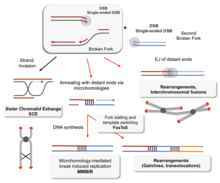
Առանց պատշաճ հոմոլոգ ռեկոմբինացիայի, քրոմոսոմները հաճախ սխալ են դասավորվում մեյոզի բջիջների բաժանման առաջին փուլի համար: Սա հանգեցնում է նրան, որ քրոմոսոմները չեն կարողանում պատշաճ կերպով տարանջատվել մի գործընթացում, որը կոչվում է ոչ տարանջատում: Իր հերթին, չտարանջատումը կարող է հանգեցնել սերմնահեղուկի և ձվաբջիջների չափազանց քիչ կամ շատ քրոմոսոմների: Դաունի համախտանիշը, որն առաջանում է 21-րդ քրոմոսոմի լրացուցիչ պատճենից, շատ աննոմալիաներից մեկն է, որոնք առաջանում են մեյոզում հոմոլոգ ռեկոմբինացիայի նման ձախողման հետևանքով.[78][103]:
Հոմոլոգ ռեկոմբինացիայի թերությունները խիստ կապված են մարդկանց մոտ քաղցկեղի առաջացման հետ: Օրինակ, քաղցկեղի հետ կապված հիվանդություններից յուրաքանչյուրը Բլումի համախտանիշը, Վերների համախտանիշը և Ռոթմունդ-Թոմսոնի համախտանիշը պայմանավորված են RecQ հելիկազի գեների անսարքությամբ, որոնք ներգրավված են հոմոլոգ ռեկոմբինացիայի կարգավորման մեջ՝ համապատասխանաբար BLM, WRN և RECQL4[104]: Բլումի սինդրոմով հիվանդների բջիջներում, որոնց բացակայում է BLM սպիտակուցի աշխատանքային օրինակը, կա հոմոլոգ ռեկոմբինացիայի բարձր մակարդակ[105]: BLM-ի անբավարարություն ունեցող մկների վրա իրականացված փորձերը ցույց են տվել, որ մուտացիան առաջացնում է քաղցկեղ՝ հետերոզիգոտության կորստի պատճառով, որն առաջանում է հոմոլոգ ռեկոմբինացիայի ավելացման հետևանքով[106]: Հետերոզիգոտության կորուստը վերաբերում է գենի երկու տարբերակներից մեկի կամ ալելների կորստին: Եթե կորցրած ալելներից մեկն օգնում է ճնշել ուռուցքները, ինչպես օրինակ ռետինոբլաստոմայի սպիտակուցի գենը, ապա հետերոզիգոտության կորուստը կարող է հանգեցնել քաղցկեղի[107]:
Հոմոլոգ ռեկոմբինացիայի արագության նվազումը հանգեցնում է ԴՆԹ-ի անարդյունավետ վերականգնման[107], որը կարող է նաև հանգեցնել քաղցկեղի[108]: Դա վերաբերում է BRCA1-ին և BRCA2-ին՝ երկու նմանատիպ ուռուցքային ճնշող գեներին, որոնց անսարքությունը կապված է կրծքագեղձի և ձվարանների քաղցկեղի առաջացման ռիսկի զգալիորեն մեծացման հետ: BRCA1 և BRCA2 բացակայող բջիջները ունեն հոմոլոգ ռեկոմբինացիայի նվազում և իոնացնող ճառագայթման նկատմամբ զգայունության բարձրացում, ինչը ենթադրում է, որ հոմոլոգ ռեկոմբինացիայի նվազումը հանգեցնում է քաղցկեղի նկատմամբ զգայունության բարձրացման[108]: Քանի որ BRCA2-ի միակ հայտնի գործառույթը հոմոլոգ ռեկոմբինացիա սկսելն է, հետազոտողները ենթադրել են, որ BRCA2-ի դերի ավելի մանրամասն իմացությունը հոմոլոգ ռեկոմբինացիայի մեջ կարող է լինել կրծքագեղձի և ձվարանների քաղցկեղի պատճառները հասկանալու բանալին[108]:
Հոմոլոգ ռեկոմբինացիայի անբավարարությամբ ուռուցքները (ներառյալ BRCA արատները) նկարագրվում են որպես հոմոլոգ ռեկոմբինացիայով օժտված-դրական[109]:
Էվոլյուցիոն պահպանություն
[խմբագրել | խմբագրել կոդը]
Թեև ուղիները կարող են մեխանիկորեն տարբերվել, օրգանիզմների հոմոլոգ ռեկոմբինացում կատարելու ունակությունը համընդհանուր պահպանված է կյանքի բոլոր ոլորտներում[110]: Հիմնվելով նրանց ամինաթթուների հաջորդականությունների նմանության վրա՝ մի շարք սպիտակուցների հոմոլոգներ կարելի է գտնել կյանքի բազմաթիվ տիրույթներում, ինչը ցույց է տալիս, որ դրանք վաղուց են զարգացել և այդ ժամանակվանից շեղվել են ընդհանուր նախնիների սպիտակուցներից[110]:
RecA ռեկոմբինազի ընտանիքի անդամները հայտնաբերված են գրեթե բոլոր օրգանիզմներում RecA-ով բակտերիաներում, Rad51 և DMC1՝ էուկարիոտներում, RadA՝ արքեայում և UvsX՝ T4 ֆագում[111]:
Հարակից միաշղթա կապող սպիտակուցներ, որոնք կարևոր են հոմոլոգ ռեկոմբինացիայի և շատ այլ գործընթացների համար, նույնպես հայտնաբերված են կյանքի բոլոր տիրույթներում[112]:
Rad54, Mre11, Rad50 և մի շարք այլ սպիտակուցներ նույնպես հայտնաբերվել են ինչպես արքեայում, այնպես էլ էուկարիոտներում[110][111][113]:
RecA ռեկոմբինազ ընտանիք
[խմբագրել | խմբագրել կոդը]Ենթադրվում է, որ RecA ռեկոմբինազների ընտանիքի սպիտակուցները սերում են ընդհանուր նախնի ռեկոմբինազից[110]: RecA ռեկոմբինազների ընտանիքը պարունակում է RecA սպիտակուցներ բակտերիայից, Rad51 և Dmc1 սպիտակուցներ էուկարիոտներից և RadA՝ արքեայից և ռեկոմբինազ պարալոգ սպիտակուցներ: Rad51, Dmc1 և RadA սպիտակուցների միջև էվոլյուցիոն հարաբերությունները մոդելավորող ուսումնասիրությունները ցույց են տալիս, որ դրանք մոնոֆիլետիկ են կամ ունեն ընդհանուր մոլեկուլային նախահայր[110]: Այս սպիտակուցային ընտանիքում Rad51-ը և Dmc1-ը խմբավորված են RadA-ից առանձին կլադի մեջ: Այս երեք սպիտակուցները միասին խմբավորելու պատճառներից մեկն այն է, որ նրանք բոլորն ունեն փոփոխված պարույր-շրջադարձ-պարույր մոտիվ, որն օգնում է սպիտակուցներին միանալ ԴՆԹ-ին՝ դեպի իրենց N-տերմինալ ծայրերը[110]: Որպես ժամանակակից RAD51 և DMC1 գեների հավանական ծագում առաջարկվել է էուկարիոտիկ RecA գենի գեների կրկնօրինակման հնագույն իրադարձություն և դրան հաջորդող մուտացիա[110]։
Սպիտակուցներն ընդհանուր առմամբ ունեն երկար պահպանված տարածք, որը հայտնի է որպես RecA/Rad51 տիրույթ: Այս սպիտակուցային տիրույթում կան երկու հաջորդական մոտիվներ՝ Walker A մոտիվը և Walker B մոտիվը: Walker A և B մոտիվները թույլ են տալիս RecA/Rad51 սպիտակուցների ընտանիքի անդամներին ներգրավվել ԱԵՖ-ի կապում և ԱԵՖ հիդրոլիզում[110][114]:
Մեյոզին հատուկ սպիտակուցներ
[խմբագրել | խմբագրել կոդը]Dmc1-ի հայտնաբերումը Giardia-ի մի քանի տեսակների մեջ, որը ամենավաղ պրոտիստներից էր, որը տարբերվում էր որպես էուկարիոտ, ենթադրում է, որ մեյոտիկ հոմոլոգ վերահամակցումը, և, հետևաբար, ինքը՝ մեյոզը, առաջացել է էուկարիոտների էվոլյուցիայի շատ վաղ շրջանում[115]: Ի լրումն Dmc1-ի վերաբերյալ հետազոտությունների, Spo11 սպիտակուցի վերաբերյալ ուսումնասիրությունները տեղեկատվություն են տրամադրել մեյոտիկ վերակոմբինացիայի ծագման մասին[116]: Spo11-ը, II տիպի տոպոիզոմերազը, կարող է մեյոզում սկսել հոմոլոգ ռեկոմբինացիա՝ ԴՆԹ-ում կրկնակի շղթայական նպատակային ընդմիջումներ կատարելով[25]։ Ֆիլոգենետիկ ծառերը, որոնք հիմնված են կենդանիների, սնկերի, բույսերի, պրոտիստների և արխեաների SPO11-ին նման գեների հաջորդականության վրա, գիտնականներին ստիպել են ենթադրել, որ ներկայումս էուկարիոտներում Spo11 տարբերակը առաջացել է էուկարիոտների և արքեաների վերջին ընդհանուր նախնուց[116]:
Տեխնոլոգիական կիրառություններ
[խմբագրել | խմբագրել կոդը]Գենային թիրախավորում
[խմբագրել | խմբագրել կոդը]
ԴՆԹ-ի հաջորդականությունները օրգանիզմներ ներմուծելու շատ մեթոդներ՝ ռեկոմբինանտ ԴՆԹ և գենետիկորեն ձևափոխված օրգանիզմներ ստեղծելու համար, օգտագործում են հոմոլոգ ռեկոմբինացիայի գործընթացը[117]։ Նաև կոչվում է գենային թիրախավորում, մեթոդը հատկապես տարածված է խմորասնկերի և մկների գենետիկայի մեջ: Գենային թիրախավորման մեթոդը նոկաուտ մկների մոտ օգտագործում է մկների սաղմնային ցողունային բջիջները՝ արհեստական գենետիկական նյութ առաքելու համար (հիմնականում բուժական հետաքրքրություն է ներկայացնում), որը ճնշում է մկան թիրախային գենը հոմոլոգ ռեկոմբինացիայի սկզբունքով: Այդպիսով մկնիկը գործում է որպես աշխատանքային մոդել՝ հասկանալու կոնկրետ կաթնասունների գենի ազդեցությունը: Ի ճանաչում իրենց հայտնագործության, թե ինչպես կարելի է հոմոլոգ ռեկոմբինացիա օգտագործել սաղմնային ցողունային բջիջների միջոցով մկների մեջ գենետիկ մոդիֆիկացիաներ ներմուծելու համար՝ Մարիո Կապեկին, Մարտին Էվանսը և Օլիվեր Սմիթիսը արժանացան 2007թվականի Նոբելյան մրցանակի ֆիզիոլոգիայի կամ բժշկության ոլորտում[118]:
Գենային թիրախավորման տեխնոլոգիաների առաջընթացը, որն խաթարում է բջիջների հոմոլոգ ռեկոմբինացման մեխանիզմը, այժմ հանգեցնում է մարդու հիվանդությունների ավելի ճշգրիտ, իզոգեն մոդելների նոր ալիքի զարգացմանը: Ենթադրվում է, որ այս մշակված մարդկային բջիջների մոդելները ավելի ճշգրիտ արտացոլում են մարդու հիվանդությունների գենետիկան, քան մկների մոդելները: Սա հիմնականում պայմանավորված է նրանով, որ հետաքրքրող մուտացիաները ներմուծվում են էնդոգեն գեներում, ճիշտ այնպես, ինչպես դրանք տեղի են ունենում իրական հիվանդների մոտ, և քանի որ դրանք հիմնված են ոչ թե առնետների, այլ մարդու գենոմի վրա: Ավելին, որոշ տեխնոլոգիաներ հնարավորություն են տալիս բախվել որոշակի մուտացիայի, այլ ոչ թե պարզապես նոկաուտների՝ կապված հին գեների թիրախավորման տեխնոլոգիաների հետ:
Սպիտակուցային ճարտարագիտություն
[խմբագրել | խմբագրել կոդը]Սպիտակուցների ճարտարագիտությունը հոմոլոգ ռեկոմբինացիայով զարգացնում է քիմերային սպիտակուցներ՝ բեկորները երկու ծնողական սպիտակուցների միջև փոխանակելով: Այս տեխնիկան օգտագործում է այն փաստը, որ ռեկոմբինացիան կարող է ներկայացնել հաջորդականության բարձր աստիճանի բազմազանություն՝ միաժամանակ պահպանելով սպիտակուցի կարողությունը ծալվելու իր երրորդական կառուցվածքի կամ եռաչափ ձևի մեջ[119]։ Սա հակադրվում է սպիտակուցների ճարտարագիտության այլ մեթոդների, ինչպիսիք են պատահական կետային մուտագենեզը, որի դեպքում սպիտակուցի ֆունկցիան պահպանելու հավանականությունը էքսպոնենցիալ նվազում է ամինաթթուների փոխարինման ավելացման հետ մեկտեղ[120]։ Ռեկոմբինացիայի տեխնիկայի միջոցով արտադրված քիմերաներն ի վիճակի են պահպանել ծալվելու իրենց կարողությունը, քանի որ նրանց փոխված ծնողական բեկորները կառուցվածքային և էվոլյուցիոն առումով պահպանված են: Այս ռեկոմբինանտ «շինանյութերը» պահպանում են կառուցվածքային կարևոր փոխազդեցությունները, ինչպիսիք են սպիտակուցի կառուցվածքում տարբեր ամինաթթուների միջև ֆիզիկական շփման կետերը: Հաշվողական մեթոդները, ինչպիսիք են SCHEMA-ն և վիճակագրական զուգավորում վերլուծությունը, կարող են օգտագործվել՝ բացահայտելու կառուցվածքային ստորաբաժանումները, որոնք հարմար են ռեկոմբինացման համար[121][122][123]:
Տեխնիկաները, որոնք հիմնված են հոմոլոգ ռեկոմբինացման վրա, օգտագործվել են նոր սպիտակուցներ մշակելու համար[121]: 2007 թվականին հրապարակված ուսումնասիրության մեջ հետազոտողները կարողացան ստեղծել երկու ֆերմենտների քիմերաներ, որոնք ներգրավված են իզոպրեոիդների կենսասինթեզում, միացությունների բազմազան դասի, այդ թվում՝ հորմոնների, տեսողական պիգմենտների և որոշ ֆերոմոնների: Քիմերային սպիտակուցները ձեռք բերեցին էական ռեակցիա կատալիզացնելու հատկություն իզոպրեոիդային կենսասինթեզի մեջ՝ բնության մեջ հայտնաբերված կենսասինթեզի ամենատարբեր ուղիներից մեկը, որը բացակայում էր մայր սպիտակուցներում[124]: Սպիտակուցների ինժեներիան ռեկոմբինացման միջոցով նաև արտադրել է քիմերային ֆերմենտներ՝ նոր ֆունկցիայով սպիտակուցների խմբի անդամների մոտ, որը հայտնի է որպես ցիտոքրոմ P450 ընտանիք[125], որը մարդկանց մոտ ներգրավված է օտար միացությունների դետոքսիկացման մեջ, ինչպիսիք են դեղամիջոցները, սննդային հավելումները և կոնսերվանտները[22]:
Քաղցկեղի բուժում
[խմբագրել | խմբագրել կոդը]Հոմոլոգ ռեկոմբինացիայով օժտված (HRP) քաղցկեղի բջիջներն ի վիճակի են վերականգնել ԴՆԹ-ի վնասը, որն առաջանում է քիմիաթերապիայի արդյունքում, ինչպիսին է ցիսպլատինը: Այսպիսով, հոմոլոգ ռեկոմբինացիայով օժտված քաղցկեղը դժվար է բուժել: Ուսումնասիրությունները ցույց են տալիս, որ հոմոլոգ ռեկոմբինացիան կարող է թիրախավորվել c-Abl արգելակման միջոցով[126][127]: BRCA մուտացիաներով քաղցկեղի բջիջներն ունեն հոմոլոգ ռեկոմբինացիայի թերություններ, և այդ թերությունները շահագործելու համար դեղամիջոցները մշակվել և հաջողությամբ օգտագործվել են կլինիկական փորձարկումներում[128][129]: Օլապարիբը՝ PARP1 ինհիբիտորը, կրճատել կամ դադարեցրել է կրծքագեղձի, ձվարանների և շագանակագեղձի քաղցկեղի ուռուցքների աճը, որոնք առաջացել են BRCA1 կամ BRCA2 գեների մուտացիաների հետևանքով, որոնք անհրաժեշտ են հոմոլոգ ռեկոմբինացիայի համար: Երբ BRCA1-ը կամ BRCA2-ը բացակայում է, ԴՆԹ-ի վերականգնող մեխանիզմների այլ տեսակներ պետք է փոխհատուցեն հոմոլոգ ռեկոմբինացիայի պակասը, ինչպես օրինակ՝ բազային հեռացման վերանորոգումը (BER) ընդհատվող վերարտադրման պատառաքաղների համար կամ ոչ հոմոլոգ ծայրի միացումը (NHEJ)՝ կրկնակի շղթայի ճեղքերի դեպքում[128]: Արգելակելով BER-ը հոմոլոգ ռեկոմբիանցիայի-դեֆիցիտի բջիջում, օլապարիբը կիրառում է սինթետիկ մահացության գաղափարը հատուկ թիրախավորված քաղցկեղային բջիջների համար: Թեև PARP1 ինհիբիտորները ներկայացնում են քաղցկեղի թերապիայի նոր մոտեցում, հետազոտողները զգուշացրել են, որ դրանք կարող են անբավարար լինել ուշ փուլի մետաստատիկ քաղցկեղը բուժելու համար[128]: Քաղցկեղի բջիջները կարող են դառնալ դիմացկուն PARP1 ինհիբիտորի նկատմամբ, եթե դրանք ենթարկվեն BRCA2 մուտացիաների ջնջման, որը խաթարում է դեղամիջոցի սինթետիկ մահացությունը՝ վերականգնելով քաղցկեղի բջիջների՝ ԴՆԹ-ն հոմոլոգ ռեկոմբինացիայով վերականգնելու կարողությունը[130]:
Տես նաև
[խմբագրել | խմբագրել կոդը]Ծանոթագրություններ
[խմբագրել | խմբագրել կոդը]- ↑ Thompson LH, Schild D (June 2001). «Homologous recombinational repair of DNA ensures mammalian chromosome stability». Mutation Research. 477 (1–2): 131–53. doi:10.1016/S0027-5107(01)00115-4. PMID 11376695.
- ↑ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P, և այլք: (2002). «Chapter 5: DNA Replication, Repair, and Recombination». Molecular Biology of the Cell (4th ed.). New York: Garland Science. էջ 845. ISBN 978-0-8153-3218-3.
- ↑ Capecchi MR (June 1989). «Altering the genome by homologous recombination». Science. 244 (4910): 1288–92. Bibcode:1989Sci...244.1288C. doi:10.1126/science.2660260. PMID 2660260.
- ↑ Smithies O, Gregg RG, Boggs SS, Koralewski MA, Kucherlapati RS (1985-09-19). «Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination». Nature. 317 (6034): 230–4. Bibcode:1985Natur.317..230S. doi:10.1038/317230a0. PMID 2995814.
- ↑ Orr-Weaver TL, Szostak JW, Rothstein RJ (October 1981). «Yeast transformation: a model system for the study of recombination». Proceedings of the National Academy of Sciences of the United States of America. 78 (10): 6354–8. Bibcode:1981PNAS...78.6354O. doi:10.1073/pnas.78.10.6354. PMC 349037. PMID 6273866.
- ↑ Orr-Weaver TL, Szostak JW (July 1983). «Yeast recombination: the association between double-strand gap repair and crossing-over». Proceedings of the National Academy of Sciences of the United States of America. 80 (14): 4417–21. Bibcode:1983PNAS...80.4417O. doi:10.1073/pnas.80.14.4417. PMC 384049. PMID 6308623.
- ↑ 7,0 7,1 Szostak JW, Orr-Weaver TL, Rothstein RJ, Stahl FW (May 1983). «The double-strand-break repair model for recombination». Cell. 33 (1): 25–35. doi:10.1016/0092-8674(83)90331-8. PMID 6380756.
- ↑ Resnick MA (June 1976). «The repair of double-strand breaks in DNA; a model involving recombination». Journal of Theoretical Biology. 59 (1): 97–106. Bibcode:1976JThBi..59...97R. doi:10.1016/s0022-5193(76)80025-2. PMID 940351.
- ↑ Jasin M, Rothstein R (November 2013). «Repair of strand breaks by homologous recombination». Cold Spring Harbor Perspectives in Biology. 5 (11): a012740. doi:10.1101/cshperspect.a012740. PMC 3809576. PMID 24097900.
- ↑ Bateson P (August 2002). «William Bateson: a biologist ahead of his time» (PDF). Journal of Genetics. 81 (2): 49–58. doi:10.1007/BF02715900. PMID 12532036.
- ↑ «Reginald Crundall Punnett». NAHSTE, University of Edinburgh. Վերցված է 3 July 2010-ին.
- ↑ Lobo I, Shaw K (2008). «Thomas Hunt Morgan, genetic recombination, and gene mapping». Nature Education. 1 (1).
- ↑ 13,0 13,1 Coe E, Kass LB (May 2005). «Proof of physical exchange of genes on the chromosomes». Proceedings of the National Academy of Sciences of the United States of America. 102 (19): 6641–6. Bibcode:2005PNAS..102.6641C. doi:10.1073/pnas.0407340102. PMC 1100733. PMID 15867161.
- ↑ Creighton HB, McClintock B (August 1931). «A Correlation of Cytological and Genetical Crossing-Over in Zea Mays». Proceedings of the National Academy of Sciences of the United States of America. 17 (8): 492–7. Bibcode:1931PNAS...17..492C. doi:10.1073/pnas.17.8.492. PMC 1076098. PMID 16587654.
- ↑ Stern, C (1931). «Zytologisch-genetische untersuchungen alsbeweise fur die Morgansche theorie des faktoraustauschs». Biologisches Zentralblatt. 51: 547–587.
- ↑ «The development of bacterial genetics». US National Library of Medicine. Վերցված է 3 July 2010-ին.
- ↑ «The Nobel Prize in Physiology or Medicine 1958». Nobelprize.org. Վերցված է 3 July 2010-ին.
- ↑ 18,0 18,1 18,2 Haber JE, Ira G, Malkova A, Sugawara N (January 2004). «Repairing a double-strand chromosome break by homologous recombination: revisiting Robin Holliday's model». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 359 (1441): 79–86. doi:10.1098/rstb.2003.1367. PMC 1693306. PMID 15065659.
- ↑ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). «12.5: Recombination between Homologous DNA Sites: Double-Strand Breaks in DNA Initiate Recombination». Molecular Cell Biology (4th ed.). W. H. Freeman and Company. ISBN 978-0-7167-3136-8.
- ↑ Griffiths A, և այլք: (1999). «8: Chromosome Mutations: Chromosomal Rearrangements». Modern Genetic Analysis. W. H. Freeman and Company. ISBN 978-0-7167-3118-4.
- ↑ Khanna KK, Jackson SP (March 2001). «DNA double-strand breaks: signaling, repair and the cancer connection». Nature Genetics. 27 (3): 247–54. doi:10.1038/85798. PMID 11242102.
- ↑ 22,0 22,1 22,2 Nelson DL, Cox MM (2005). Principles of Biochemistry (4th ed.). Freeman. էջեր 980–981. ISBN 978-0-7167-4339-2.
- ↑ Marcon E, Moens PB (August 2005). «The evolution of meiosis: recruitment and modification of somatic DNA-repair proteins». BioEssays. 27 (8): 795–808. doi:10.1002/bies.20264. PMID 16015600.
- ↑ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. էջ 305. ISBN 978-0-8153-4105-5.
- ↑ 25,0 25,1 Keeney S, Giroux CN, Kleckner N (February 1997). «Meiosis-specific DNA double-strand breaks are catalyzed by Spo11, a member of a widely conserved protein family». Cell. 88 (3): 375–84. doi:10.1016/S0092-8674(00)81876-0. PMID 9039264.
- ↑ Longhese MP, Bonetti D, Guerini I, Manfrini N, Clerici M (September 2009). «DNA double-strand breaks in meiosis: checking their formation, processing and repair». DNA Repair. 8 (9): 1127–38. doi:10.1016/j.dnarep.2009.04.005. PMID 19464965.
- ↑ Cahill LP, Mariana JC, Mauléon P (January 1979). «Total follicular populations in ewes of high and low ovulation rates». Journal of Reproduction and Fertility. 55 (1): 27–36. doi:10.1530/jrf.0.0550027. PMID 423159.
- ↑ Schimmel J, van Schendel R, den Dunnen JT, Tijsterman M (September 2019). «Templated Insertions: A Smoking Gun for Polymerase Theta-Mediated End Joining». Trends in Genetics (անգլերեն). 35 (9): 632–644. doi:10.1016/j.tig.2019.06.001. PMID 31296341.
- ↑ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. էջ 303. ISBN 978-0-8153-4105-5.
- ↑ 30,0 30,1 Shrivastav M, De Haro LP, Nickoloff JA (January 2008). «Regulation of DNA double-strand break repair pathway choice». Cell Research. 18 (1): 134–47. doi:10.1038/cr.2007.111. PMID 18157161.
- ↑ 31,0 31,1 Mimitou EP, Symington LS (May 2009). «Nucleases and helicases take center stage in homologous recombination». Trends in Biochemical Sciences. 34 (5): 264–72. doi:10.1016/j.tibs.2009.01.010. PMID 19375328.
- ↑ Andres, Sara N.; Williams, R. Scott (August 2017). «CtIP/Ctp1/Sae2, molecular form fit for function». DNA Repair. 56: 109–117. doi:10.1016/j.dnarep.2017.06.013. PMC 5543718. PMID 28623092.
- ↑ Huertas P, Cortés-Ledesma F, Sartori AA, Aguilera A, Jackson SP (October 2008). «CDK targets Sae2 to control DNA-end resection and homologous recombination». Nature. 455 (7213): 689–92. Bibcode:2008Natur.455..689H. doi:10.1038/nature07215. PMC 2635538. PMID 18716619.
- ↑ Liu B, Yip RK, Zhou Z (2012). «Chromatin remodeling, DNA damage repair and aging». Curr. Genomics. 13 (7): 533–47. doi:10.2174/138920212803251373. PMC 3468886. PMID 23633913.
- ↑ 35,0 35,1 35,2 35,3 Sellou H, Lebeaupin T, Chapuis C, Smith R, Hegele A, Singh HR, Kozlowski M, Bultmann S, Ladurner AG, Timinszky G, Huet S (2016). «The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage». Mol. Biol. Cell. 27 (24): 3791–3799. doi:10.1091/mbc.E16-05-0269. PMC 5170603. PMID 27733626.
- ↑ 36,0 36,1 Van Meter M, Simon M, Tombline G, May A, Morello TD, Hubbard BP, Bredbenner K, Park R, Sinclair DA, Bohr VA, Gorbunova V, Seluanov A (2016). «JNK Phosphorylates SIRT6 to Stimulate DNA Double-Strand Break Repair in Response to Oxidative Stress by Recruiting PARP1 to DNA Breaks». Cell Rep. 16 (10): 2641–50. doi:10.1016/j.celrep.2016.08.006. PMC 5089070. PMID 27568560.
- ↑ 37,0 37,1 Haince JF, McDonald D, Rodrigue A, Déry U, Masson JY, Hendzel MJ, Poirier GG (2008). «PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites». J. Biol. Chem. 283 (2): 1197–208. doi:10.1074/jbc.M706734200. PMID 18025084.
- ↑ 38,0 38,1 38,2 Rogakou EP, Pilch DR, Orr AH, Ivanova VS, Bonner WM (1998). «DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139». J. Biol. Chem. 273 (10): 5858–68. doi:10.1074/jbc.273.10.5858. PMID 9488723.
- ↑ Mailand N, Bekker-Jensen S, Faustrup H, Melander F, Bartek J, Lukas C, Lukas J (2007). «RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins». Cell. 131 (5): 887–900. doi:10.1016/j.cell.2007.09.040. PMID 18001824.
- ↑ Luijsterburg MS, Acs K, Ackermann L, Wiegant WW, Bekker-Jensen S, Larsen DH, Khanna KK, van Attikum H, Mailand N, Dantuma NP (2012). «A new non-catalytic role for ubiquitin ligase RNF8 in unfolding higher-order chromatin structure». EMBO J. 31 (11): 2511–27. doi:10.1038/emboj.2012.104. PMC 3365417. PMID 22531782.
- ↑ Baudat F, Buard J, Grey C, Fledel-Alon A, Ober C, Przeworski M, և այլք: (February 2010). «PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice». Science. 327 (5967): 836–40. Bibcode:2010Sci...327..836B. doi:10.1126/science.1183439. PMC 4295902. PMID 20044539.
- ↑ Wells D, Bitoun E, Moralli D, Zhang G, Hinch A, Jankowska J, և այլք: (August 2020). «ZCWPW1 is recruited to recombination hotspots by PRDM9, and is essential for meiotic double strand break repair». eLife. 9: e53392. doi:10.7554/eLife.53392. PMC 7494361. PMID 32744506.
- ↑ 43,0 43,1 43,2 43,3 Sung P, Klein H (October 2006). «Mechanism of homologous recombination: mediators and helicases take on regulatory functions». Nature Reviews Molecular Cell Biology. 7 (10): 739–50. doi:10.1038/nrm2008. PMID 16926856.
- ↑ Wold MS (1997). «Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism». Annual Review of Biochemistry. 66: 61–92. doi:10.1146/annurev.biochem.66.1.61. PMID 9242902.
- ↑ McMahill MS, Sham CW, Bishop DK (November 2007). «Synthesis-dependent strand annealing in meiosis». PLOS Biology. 5 (11): e299. doi:10.1371/journal.pbio.0050299. PMC 2062477. PMID 17988174.

- ↑ Bärtsch S, Kang LE, Symington LS (February 2000). «RAD51 is required for the repair of plasmid double-stranded DNA gaps from either plasmid or chromosomal templates». Molecular and Cellular Biology. 20 (4): 1194–205. doi:10.1128/MCB.20.4.1194-1205.2000. PMC 85244. PMID 10648605.
- ↑ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. էջեր 312–313. ISBN 978-0-8153-4105-5.
- ↑ 48,0 48,1 48,2 Helleday T, Lo J, van Gent DC, Engelward BP (July 2007). «DNA double-strand break repair: from mechanistic understanding to cancer treatment». DNA Repair. 6 (7): 923–35. doi:10.1016/j.dnarep.2007.02.006. PMID 17363343.
- ↑ 49,0 49,1 Andersen SL, Sekelsky J (December 2010). «Meiotic versus mitotic recombination: two different routes for double-strand break repair: the different functions of meiotic versus mitotic DSB repair are reflected in different pathway usage and different outcomes». BioEssays. 32 (12): 1058–66. doi:10.1002/bies.201000087. PMC 3090628. PMID 20967781.
- ↑ Allers T, Lichten M (July 2001). «Differential timing and control of noncrossover and crossover recombination during meiosis». Cell. 106 (1): 47–57. doi:10.1016/s0092-8674(01)00416-0. PMID 11461701.
- ↑ Haber lab. «Single-strand annealing». Brandeis University. Արխիվացված է օրիգինալից 19 January 2015-ին. Վերցված է 3 July 2010-ին.
- ↑ 52,0 52,1 52,2 Lyndaker AM, Alani E (March 2009). «A tale of tails: insights into the coordination of 3' end processing during homologous recombination». BioEssays. 31 (3): 315–21. doi:10.1002/bies.200800195. PMC 2958051. PMID 19260026.
- ↑ Mimitou EP, Symington LS (September 2009). «DNA end resection: many nucleases make light work». DNA Repair. 8 (9): 983–95. doi:10.1016/j.dnarep.2009.04.017. PMC 2760233. PMID 19473888.
- ↑ Pâques F, Haber JE (June 1999). «Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae». Microbiology and Molecular Biology Reviews. 63 (2): 349–404. doi:10.1128/MMBR.63.2.349-404.1999. PMC 98970. PMID 10357855.
- ↑ 55,0 55,1 McEachern MJ, Haber JE (2006). «Break-induced replication and recombinational telomere elongation in yeast». Annual Review of Biochemistry. 75: 111–35. doi:10.1146/annurev.biochem.74.082803.133234. PMID 16756487.
- ↑ Morrish TA, Greider CW (January 2009). Haber JE (ed.). «Short telomeres initiate telomere recombination in primary and tumor cells». PLOS Genetics. 5 (1): e1000357. doi:10.1371/journal.pgen.1000357. PMC 2627939. PMID 19180191.

- ↑ Muntoni A, Reddel RR (October 2005). «The first molecular details of ALT in human tumor cells». Human Molecular Genetics. 14 Spec No. 2 (Review Issue 2): R191–6. doi:10.1093/hmg/ddi266. PMID 16244317.
- ↑ Chen Z, Yang H, Pavletich NP (May 2008). «Mechanism of homologous recombination from the RecA-ssDNA/dsDNA structures». Nature. 453 (7194): 489–4. Bibcode:2008Natur.453..489C. doi:10.1038/nature06971. PMID 18497818.
- ↑ Kowalczykowski SC, Dixon DA, Eggleston AK, Lauder SD, Rehrauer WM (September 1994). «Biochemistry of homologous recombination in Escherichia coli». Microbiological Reviews. 58 (3): 401–65. doi:10.1128/MMBR.58.3.401-465.1994. PMC 372975. PMID 7968921.
- ↑ Rocha EP, Cornet E, Michel B (August 2005). «Comparative and evolutionary analysis of the bacterial homologous recombination systems». PLOS Genetics. 1 (2): e15. doi:10.1371/journal.pgen.0010015. PMC 1193525. PMID 16132081.

- ↑ 61,0 61,1 Amundsen SK, Taylor AF, Reddy M, Smith GR (December 2007). «Intersubunit signaling in RecBCD enzyme, a complex protein machine regulated by Chi hot spots». Genes & Development. 21 (24): 3296–307. doi:10.1101/gad.1605807. PMC 2113030. PMID 18079176.
- ↑ Singleton MR, Dillingham MS, Gaudier M, Kowalczykowski SC, Wigley DB (November 2004). «Crystal structure of RecBCD enzyme reveals a machine for processing DNA breaks» (PDF). Nature. 432 (7014): 187–93. Bibcode:2004Natur.432..187S. doi:10.1038/nature02988. PMID 15538360. Արխիվացված է օրիգինալից (PDF) 2004-05-25-ին.
- ↑ Cromie GA (August 2009). «Phylogenetic ubiquity and shuffling of the bacterial RecBCD and AddAB recombination complexes». Journal of Bacteriology. 191 (16): 5076–84. doi:10.1128/JB.00254-09. PMC 2725590. PMID 19542287.
- ↑ 64,0 64,1 Smith GR (June 2012). «How RecBCD enzyme and Chi promote DNA break repair and recombination: a molecular biologist's view». Microbiology and Molecular Biology Reviews. 76 (2): 217–28. doi:10.1128/MMBR.05026-11. PMC 3372252. PMID 22688812.
- ↑ 65,0 65,1 65,2 65,3 Dillingham MS, Kowalczykowski SC (December 2008). «RecBCD enzyme and the repair of double-stranded DNA breaks». Microbiology and Molecular Biology Reviews. 72 (4): 642–71, Table of Contents. doi:10.1128/MMBR.00020-08. PMC 2593567. PMID 19052323.
- ↑ Michel B, Boubakri H, Baharoglu Z, LeMasson M, Lestini R (July 2007). «Recombination proteins and rescue of arrested replication forks». DNA Repair. 6 (7): 967–80. doi:10.1016/j.dnarep.2007.02.016. PMID 17395553.
- ↑ Spies M, Bianco PR, Dillingham MS, Handa N, Baskin RJ, Kowalczykowski SC (September 2003). «A molecular throttle: the recombination hotspot chi controls DNA translocation by the RecBCD helicase». Cell. 114 (5): 647–54. doi:10.1016/S0092-8674(03)00681-0. PMID 13678587.
- ↑ Taylor AF, Smith GR (June 2003). «RecBCD enzyme is a DNA helicase with fast and slow motors of opposite polarity». Nature. 423 (6942): 889–93. Bibcode:2003Natur.423..889T. doi:10.1038/nature01674. PMID 12815437.
- ↑ Spies M, Amitani I, Baskin RJ, Kowalczykowski SC (November 2007). «RecBCD enzyme switches lead motor subunits in response to chi recognition». Cell. 131 (4): 694–705. doi:10.1016/j.cell.2007.09.023. PMC 2151923. PMID 18022364.
- ↑ Savir Y, Tlusty T (November 2010). «RecA-mediated homology search as a nearly optimal signal detection system» (PDF). Molecular Cell. 40 (3): 388–96. Bibcode:2010arXiv1011.4382S. doi:10.1016/j.molcel.2010.10.020. PMID 21070965. Արխիվացված է օրիգինալից (PDF) 2012-10-07-ին. Վերցված է 2011-08-31-ին.
- ↑ Rambo RP, Williams GJ, Tainer JA (November 2010). «Achieving fidelity in homologous recombination despite extreme complexity: informed decisions by molecular profiling» (PDF). Molecular Cell. 40 (3): 347–8. doi:10.1016/j.molcel.2010.10.032. PMC 3003302. PMID 21070960. Արխիվացված է օրիգինալից (PDF) 2012-10-07-ին. Վերցված է 2011-08-31-ին.
- ↑ De Vlaminck I, van Loenhout MT, Zweifel L, den Blanken J, Hooning K, Hage S, և այլք: (June 2012). «Mechanism of homology recognition in DNA recombination from dual-molecule experiments». Molecular Cell. 46 (5): 616–24. doi:10.1016/j.molcel.2012.03.029. PMID 22560720.
- ↑ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. էջ 307. ISBN 978-0-8153-4105-5.
- ↑ Morimatsu K, Kowalczykowski SC (May 2003). «RecFOR proteins load RecA protein onto gapped DNA to accelerate DNA strand exchange: a universal step of recombinational repair». Molecular Cell. 11 (5): 1337–47. doi:10.1016/S1097-2765(03)00188-6. PMID 12769856.
- ↑ Hiom K (July 2009). «DNA repair: common approaches to fixing double-strand breaks». Current Biology. 19 (13): R523–5. doi:10.1016/j.cub.2009.06.009. PMID 19602417.
- ↑ Handa N, Morimatsu K, Lovett ST, Kowalczykowski SC (May 2009). «Reconstitution of initial steps of dsDNA break repair by the RecF pathway of E. coli». Genes & Development. 23 (10): 1234–45. doi:10.1101/gad.1780709. PMC 2685532. PMID 19451222.
- ↑ 77,0 77,1 West SC (June 2003). «Molecular views of recombination proteins and their control». Nature Reviews Molecular Cell Biology. 4 (6): 435–45. doi:10.1038/nrm1127. PMID 12778123.
- ↑ 78,0 78,1 78,2 78,3 Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2003). Molecular Biology of the Gene (5th ed.). Pearson/Benjamin Cummings. էջեր 259–291. ISBN 978-0-8053-4635-0.
- ↑ Gumbiner-Russo LM, Rosenberg SM (28 November 2007). Sandler S (ed.). «Physical analyses of E. coli heteroduplex recombination products in vivo: on the prevalence of 5' and 3' patches». PLOS ONE. 2 (11): e1242. Bibcode:2007PLoSO...2.1242G. doi:10.1371/journal.pone.0001242. PMC 2082072. PMID 18043749.

- ↑ Thomas CM, Nielsen KM (September 2005). «Mechanisms of, and barriers to, horizontal gene transfer between bacteria» (PDF). Nature Reviews. Microbiology. 3 (9): 711–21. doi:10.1038/nrmicro1234. PMID 16138099. Արխիվացված է օրիգինալից (PDF) 2010-06-01-ին.
- ↑ Vulić M, Dionisio F, Taddei F, Radman M (September 1997). «Molecular keys to speciation: DNA polymorphism and the control of genetic exchange in enterobacteria». Proceedings of the National Academy of Sciences of the United States of America. 94 (18): 9763–7. Bibcode:1997PNAS...94.9763V. doi:10.1073/pnas.94.18.9763. PMC 23264. PMID 9275198.
- ↑ Majewski J, Cohan FM (January 1998). «The effect of mismatch repair and heteroduplex formation on sexual isolation in Bacillus». Genetics. 148 (1): 13–8. doi:10.1093/genetics/148.1.13. PMC 1459767. PMID 9475717.
- ↑ Majewski J, Zawadzki P, Pickerill P, Cohan FM, Dowson CG (February 2000). «Barriers to genetic exchange between bacterial species: Streptococcus pneumoniae transformation». Journal of Bacteriology. 182 (4): 1016–23. doi:10.1128/JB.182.4.1016-1023.2000. PMC 94378. PMID 10648528.
- ↑ Chen I, Dubnau D (March 2004). «DNA uptake during bacterial transformation». Nature Reviews. Microbiology. 2 (3): 241–9. doi:10.1038/nrmicro844. PMID 15083159.
- ↑ Claverys JP, Martin B, Polard P (May 2009). «The genetic transformation machinery: composition, localization, and mechanism». FEMS Microbiology Reviews. 33 (3): 643–56. doi:10.1111/j.1574-6976.2009.00164.x. PMID 19228200.
- ↑ Kidane D, Graumann PL (July 2005). «Intracellular protein and DNA dynamics in competent Bacillus subtilis cells». Cell. 122 (1): 73–84. doi:10.1016/j.cell.2005.04.036. PMID 16009134.
- ↑ Fleischmann Jr WR (1996). «43». Medical Microbiology (4th ed.). University of Texas Medical Branch at Galveston. ISBN 978-0-9631172-1-2.
- ↑ 88,0 88,1 88,2 Nagy PD, Bujarski JJ (January 1996). «Homologous RNA recombination in brome mosaic virus: AU-rich sequences decrease the accuracy of crossovers». Journal of Virology. 70 (1): 415–26. doi:10.1128/JVI.70.1.415-426.1996. PMC 189831. PMID 8523555.
- ↑ Boni MF, de Jong MD, van Doorn HR, Holmes EC (3 May 2010). Martin DP (ed.). «Guidelines for identifying homologous recombination events in influenza A virus». PLOS ONE. 5 (5): e10434. Bibcode:2010PLoSO...510434B. doi:10.1371/journal.pone.0010434. PMC 2862710. PMID 20454662.

- ↑ Chetverin AB (October 1999). «The puzzle of RNA recombination». FEBS Letters. 460 (1): 1–5. doi:10.1016/S0014-5793(99)01282-X. PMC 7163957. PMID 10571050.
- ↑ 91,0 91,1 Roossinck MJ (September 1997). «Mechanisms of plant virus evolution». Annual Review of Phytopathology. 35: 191–209. doi:10.1146/annurev.phyto.35.1.191. PMID 15012521.
- ↑ Arbuckle JH, Medveczky PG (August 2011). «The molecular biology of human herpesvirus-6 latency and telomere integration». Microbes and Infection / Institut Pasteur. 13 (8–9): 731–41. doi:10.1016/j.micinf.2011.03.006. PMC 3130849. PMID 21458587.
- ↑ Bernstein C (March 1981). «Deoxyribonucleic acid repair in bacteriophage». Microbiological Reviews. 45 (1): 72–98. doi:10.1128/MMBR.45.1.72-98.1981. PMC 281499. PMID 6261109.
- ↑ Bernstein C, Bernstein H (2001). DNA repair in bacteriophage. In: Nickoloff JA, Hoekstra MF (Eds.) DNA Damage and Repair, Vol.3. Advances from Phage to Humans. Humana Press, Totowa, NJ, pp. 1–19. 978-0896038035
- ↑ Story RM, Bishop DK, Kleckner N, Steitz TA (March 1993). «Structural relationship of bacterial RecA proteins to recombination proteins from bacteriophage T4 and yeast». Science. 259 (5103): 1892–6. Bibcode:1993Sci...259.1892S. doi:10.1126/science.8456313. PMID 8456313.
- ↑ Michod RE, Bernstein H, Nedelcu AM (May 2008). «Adaptive value of sex in microbial pathogens». Infection, Genetics and Evolution. 8 (3): 267–85. doi:10.1016/j.meegid.2008.01.002. PMID 18295550.http://www.hummingbirds.arizona.edu/Faculty/Michod/Downloads/IGE%20review%20sex.pdf Արխիվացված 2020-05-11 Wayback Machine
- ↑ 97,0 97,1 Su, Shuo; Wong, Gary; Shi, Weifeng; Liu, Jun; Lai, Alexander C.K.; Zhou, Jiyong; Liu, Wenjun; Bi, Yuhai; Gao, George F. (2016). «Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses». Trends in Microbiology. 24 (6): 490–502. doi:10.1016/j.tim.2016.03.003. PMC 7125511. PMID 27012512.
- ↑ Barr, J. N.; Fearns, R. (2010). «How RNA viruses maintain their genome integrity». Journal of General Virology. 91 (6): 1373–1387. doi:10.1099/vir.0.020818-0. PMID 20335491.
- ↑ 99,0 99,1 Li, Xiaojun; Giorgi, Elena E.; Marichannegowda, Manukumar Honnayakanahalli; Foley, Brian; Xiao, Chuan; Kong, Xiang-Peng; Chen, Yue; Gnanakaran, S.; Korber, Bette; Gao, Feng (2020). «Emergence of SARS-CoV-2 through recombination and strong purifying selection». Science Advances. 6 (27). Bibcode:2020SciA....6.9153L. doi:10.1126/sciadv.abb9153. PMC 7458444. PMID 32937441.
- ↑ Rehman, Saif ur; Shafique, Laiba; Ihsan, Awais; Liu, Qingyou (2020). «Evolutionary Trajectory for the Emergence of Novel Coronavirus SARS-CoV-2». Pathogens. 9 (3): 240. doi:10.3390/pathogens9030240. PMC 7157669. PMID 32210130.
- ↑ Yeh TY, Contreras GP (July 2020). «Emerging viral mutants in Australia suggest RNA recombination event in the SARS-CoV-2 genome». The Medical Journal of Australia. 213 (1): 44–44.e1. doi:10.5694/mja2.50657. PMC 7300921. PMID 32506536.
- ↑ Yeh TY, Contreras GP (1 July 2021). «Viral transmission and evolution dynamics of SARS-CoV-2 in shipboard quarantine». Bull. World Health Organ. 99 (7): 486–495. doi:10.2471/BLT.20.255752. PMC 8243027. PMID 34248221.
- ↑ Lamb NE, Yu K, Shaffer J, Feingold E, Sherman SL (January 2005). «Association between maternal age and meiotic recombination for trisomy 21». American Journal of Human Genetics. 76 (1): 91–9. doi:10.1086/427266. PMC 1196437. PMID 15551222.
- ↑ Cold Spring Harbor Laboratory (2007). «Human RecQ Helicases, Homologous Recombination And Genomic Instability». ScienceDaily. Վերցված է 3 July 2010-ին.
- ↑ Modesti M, Kanaar R (2001). «Homologous recombination: from model organisms to human disease». Genome Biology. 2 (5): REVIEWS1014. doi:10.1186/gb-2001-2-5-reviews1014. PMC 138934. PMID 11387040.
- ↑ Luo G, Santoro IM, McDaniel LD, Nishijima I, Mills M, Youssoufian H, Vogel H, Schultz RA, Bradley A (December 2000). «Cancer predisposition caused by elevated mitotic recombination in Bloom mice». Nature Genetics. 26 (4): 424–9. doi:10.1038/82548. PMID 11101838.
- ↑ 107,0 107,1 Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2007). Molecular Biology of the Cell (5th ed.). Garland Science. ISBN 978-0-8153-4110-9.
- ↑ 108,0 108,1 108,2 Powell SN, Kachnic LA (September 2003). «Roles of BRCA1 and BRCA2 in homologous recombination, DNA replication fidelity and the cellular response to ionizing radiation». Oncogene. 22 (37): 5784–91. doi:10.1038/sj.onc.1206678. PMID 12947386.
- ↑ «Use of homologous recombination deficiency (HRD) score to enrich for niraparib sensitive high grade ovarian tumors». Արխիվացված է օրիգինալից 2017-04-30-ին. Վերցված է 2016-12-30-ին.
- ↑ 110,0 110,1 110,2 110,3 110,4 110,5 110,6 110,7 Lin Z, Kong H, Nei M, Ma H (July 2006). «Origins and evolution of the recA/RAD51 gene family: evidence for ancient gene duplication and endosymbiotic gene transfer». Proceedings of the National Academy of Sciences of the United States of America. 103 (27): 10328–33. Bibcode:2006PNAS..10310328L. doi:10.1073/pnas.0604232103. PMC 1502457. PMID 16798872.
- ↑ 111,0 111,1 Haseltine CA, Kowalczykowski SC (May 2009). «An archaeal Rad54 protein remodels DNA and stimulates DNA strand exchange by RadA». Nucleic Acids Research. 37 (8): 2757–70. doi:10.1093/nar/gkp068. PMC 2677860. PMID 19282450.
- ↑ Rolfsmeier ML, Haseltine CA (March 2010). «The single-stranded DNA binding protein of Sulfolobus solfataricus acts in the presynaptic step of homologous recombination». Journal of Molecular Biology. 397 (1): 31–45. doi:10.1016/j.jmb.2010.01.004. PMID 20080104.
- ↑ Huang Q, Liu L, Liu J, Ni J, She Q, Shen Y (2015). «Efficient 5'-3' DNA end resection by HerA and NurA is essential for cell viability in the crenarchaeon Sulfolobus islandicus». BMC Molecular Biology. 16: 2. doi:10.1186/s12867-015-0030-z. PMC 4351679. PMID 25880130.

- ↑ Jain SK, Cox MM, Inman RB (August 1994). «On the role of ATP hydrolysis in RecA protein-mediated DNA strand exchange. III. Unidirectional branch migration and extensive hybrid DNA formation». The Journal of Biological Chemistry. 269 (32): 20653–61. doi:10.1016/S0021-9258(17)32043-4. PMID 8051165.
- ↑ Ramesh MA, Malik SB, Logsdon JM (January 2005). «A phylogenomic inventory of meiotic genes; evidence for sex in Giardia and an early eukaryotic origin of meiosis». Current Biology. 15 (2): 185–91. doi:10.1016/j.cub.2005.01.003. PMID 15668177.
- ↑ 116,0 116,1 Malik SB, Ramesh MA, Hulstrand AM, Logsdon JM (December 2007). «Protist homologs of the meiotic Spo11 gene and topoisomerase VI reveal an evolutionary history of gene duplication and lineage-specific loss». Molecular Biology and Evolution. 24 (12): 2827–41. doi:10.1093/molbev/msm217. PMID 17921483.
- ↑ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). «Chapter 8.5: Gene Replacement and Transgenic Animals: DNA Is Transferred into Eukaryotic Cells in Various Ways». Molecular Cell Biology (4th ed.). W. H. Freeman and Company. ISBN 978-0-7167-3136-8.
- ↑ «The Nobel Prize in Physiology or Medicine 2007». The Nobel Foundation. Վերցված է December 15, 2008-ին.
- ↑ Drummond DA, Silberg JJ, Meyer MM, Wilke CO, Arnold FH (April 2005). «On the conservative nature of intragenic recombination». Proceedings of the National Academy of Sciences of the United States of America. 102 (15): 5380–5. Bibcode:2005PNAS..102.5380D. doi:10.1073/pnas.0500729102. PMC 556249. PMID 15809422.
- ↑ Bloom JD, Silberg JJ, Wilke CO, Drummond DA, Adami C, Arnold FH (January 2005). «Thermodynamic prediction of protein neutrality». Proceedings of the National Academy of Sciences of the United States of America. 102 (3): 606–11. arXiv:q-bio/0409013. Bibcode:2005PNAS..102..606B. doi:10.1073/pnas.0406744102. PMC 545518. PMID 15644440.
- ↑ 121,0 121,1 Carbone MN, Arnold FH (August 2007). «Engineering by homologous recombination: exploring sequence and function within a conserved fold». Current Opinion in Structural Biology. 17 (4): 454–9. doi:10.1016/j.sbi.2007.08.005. PMID 17884462.
- ↑ Otey CR, Landwehr M, Endelman JB, Hiraga K, Bloom JD, Arnold FH (May 2006). «Structure-guided recombination creates an artificial family of cytochromes P450». PLOS Biology. 4 (5): e112. doi:10.1371/journal.pbio.0040112. PMC 1431580. PMID 16594730.

- ↑ Socolich M, Lockless SW, Russ WP, Lee H, Gardner KH, Ranganathan R (September 2005). «Evolutionary information for specifying a protein fold». Nature. 437 (7058): 512–8. Bibcode:2005Natur.437..512S. doi:10.1038/nature03991. PMID 16177782.
- ↑ Thulasiram HV, Erickson HK, Poulter CD (April 2007). «Chimeras of two isoprenoid synthases catalyze all four coupling reactions in isoprenoid biosynthesis». Science. 316 (5821): 73–6. Bibcode:2007Sci...316...73T. doi:10.1126/science.1137786. PMID 17412950.
- ↑ Landwehr M, Carbone M, Otey CR, Li Y, Arnold FH (March 2007). «Diversification of catalytic function in a synthetic family of chimeric cytochrome p450s». Chemistry & Biology. 14 (3): 269–78. doi:10.1016/j.chembiol.2007.01.009. PMC 1991292. PMID 17379142.
- ↑ Choudhury, Ananya; Zhao, Helen; Jalali, Farid; Rashid, Shahnaz AL; Ran, Jane; Supiot, Stephane; Kiltie, Anne E.; Bristow, Robert G. (1 January 2009). «Targeting homologous recombination using imatinib results in enhanced tumor cell chemosensitivity and radiosensitivity» (PDF). Molecular Cancer Therapeutics (անգլերեն). 8 (1): 203–213. doi:10.1158/1535-7163.MCT-08-0959. ISSN 1535-7163. PMID 19139130.
- ↑ Siddiqui, Arafat; Tumiati, Manuela; Joko, Alia; Sandholm, Jouko; Roering, Pia; Aakko, Sofia; Vainionpää, Reetta; Kaipio, Katja; Huhtinen, Kaisa; Kauppi, Liisa; Tuomela, Johanna; Hietanen, Sakari (2021). «Targeting DNA Homologous Repair Proficiency With Concomitant Topoisomerase II and c-Abl Inhibition». Frontiers in Oncology. 11: 3666. doi:10.3389/fonc.2021.733700. ISSN 2234-943X. PMC 8488401. PMID 34616682.
- ↑ 128,0 128,1 128,2 Iglehart JD, Silver DP (July 2009). «Synthetic lethality--a new direction in cancer-drug development». The New England Journal of Medicine. 361 (2): 189–91. doi:10.1056/NEJMe0903044. PMID 19553640.
- ↑ Fong PC, Boss DS, Yap TA, Tutt A, Wu P, Mergui-Roelvink M, Mortimer P, Swaisland H, Lau A, O'Connor MJ, Ashworth A, Carmichael J, Kaye SB, Schellens JH, de Bono JS (July 2009). «Inhibition of poly(ADP-ribose) polymerase in tumors from BRCA mutation carriers». The New England Journal of Medicine. 361 (2): 123–34. doi:10.1056/NEJMoa0900212. PMID 19553641.
- ↑ Edwards SL, Brough R, Lord CJ, Natrajan R, Vatcheva R, Levine DA, Boyd J, Reis-Filho JS, Ashworth A (February 2008). «Resistance to therapy caused by intragenic deletion in BRCA2». Nature. 451 (7182): 1111–5. Bibcode:2008Natur.451.1111E. doi:10.1038/nature06548. PMID 18264088.
Արտաքին հղումներ
[խմբագրել | խմբագրել կոդը]| Վիքիպահեստ նախագծում կարող եք այս նյութի վերաբերյալ հավելյալ պատկերազարդում գտնել Հոմոլոգ ռեկոմբինացիա կատեգորիայում։ |
- Animations – homologous recombination: Animations showing several models of homologous recombination
- Homologous recombination: Tempy & Trun: Animation of the bacterial RecBCD pathway of homologous recombination
